Haplogroupe A
En génétique humaine, l’haplogroupe A (M91) est un haplogroupe du chromosome Y. L’haplogroupe A dont l’origine et la plus grande diversité se trouvent en Afrique est également le plus ancien des haplogroupes. Il réfère un groupe de lignages du chromosome Y qui fut la première branche issue de la racine de la phylogénie du chromosome Y des humains actuels. Les mutations qui définissent l'haplogroupe A, seraient arrivées dans les chromosomes Y de quelques-uns des premiers descendants de l'Adam chromosomique, incluant au moins un de ses fils. Noter qu'une variante archaïque apportée par hybridation définit un haplogroupe distinct dénommé A00.

| Date d'origine | 140 000 avant présent[1], 270,000 avant présent[2] ou 275,000 avant présent[3] |
|---|---|
| Place d'origine | Afrique centrale[1] |
| Ancêtre | Adam-Y chromosomique |
| Descendants | A00, A0, A1a, A1b |
| Plus hautes fréquences |
Namibie (San Tsumkwe, Nama) 60-70 % Sud-Soudan (Dinka, Shilluk, Nuer) 33 %-61,5 % |
L'haplogroupe A est pratiquement concentré dans certaines parties de l'Afrique, quoique quelques cas aient été rapportés en Asie de l'Ouest. Ce clade obtient ses plus grandes fréquences actuellement chez les San, population de chasseurs-cueilleurs d'Afrique australe, suivi de près par beaucoup de groupes nilotiques en Afrique de l'Est. Cependant, les plus vieux sous-clades de l'haplogroupe A sont exclusivement trouvés en Afrique centrale. En 2011, Fulvio Cruciani et al. ont calculé par la diversité de l'ADN du chromosome Y que le plus récent ancêtre patrilinéaire commun daterait d'environ 140 000 années[1]. Ce clade a aussi été observé à des fréquences notables dans certaines populations en Éthiopie et chez quelques groupes Pygmées en Afrique Centrale.
L'haplogroupe A est moins commun parmi les locuteurs des langues nigéro-congolaises, qui appartiennent majoritairement au clade E1b1a. On pense en général que l'haplogroupe E est originaire de l'Afrique du Nord-Est[4], et s'introduisit plus tard en Afrique de l'Ouest d'où ils se répandit il y a 5 000 ans en Afrique centrale, puis du Sud et du Sud-Est avec l'expansion bantoue[5],[6]. Selon Wood et al. (2005) et Rosa et al. (2007), de tels mouvements de population relativement récents venant d'Afrique de l'Ouest changèrent la diversité pré-existante du chromosome Y dans les populations d'Afrique centrale, australe et du Sud-est, remplaçant les haplogroupes antérieurs dans ces régions par les lignages E1b1a maintenant dominants. On peut cependant observer aujourd'hui des traces des habitants ancestraux de ces régions par la présence des haplogroupes de l'ADN-Y A-M91 et B-M60 qui sont communs dans certaines populations reliques, comme les Pygmées Mbuti de la forêt d'Ituri (en République démocratique du Congo) et les Khoïsan de l'Afrique australe[7],[8],[9].
Historique

Le premier séquençage du chromosome Y humain suggérait que la première bifurcation dans l'arbre des familles du chromosome-Y se produisit avec la mutation M91 qui sépare l'haplogroupe A de l'haplogroupe BT[10]. Cependant on reconnait maintenant que la plus ancienne bifurcation de l'arbre du chromosome-Y se trouve entre deux sous-clades déjà décrits de l'haplogroupe A, plutôt qu'entre l'haplogroupe A et l'haplogroupe BT (groupe ancestral de l'ensemble des haplogroupes de B à T). Les subclades A1b and A1a-T descendent maintenant directement de la racine de l'arbre. Ce réarrangement de l'arbre des familles du chromosome-Y conduit à considérer que les lignées classées dans l'haplogroupe A ne forment pas nécessairement un clade monophylétique[1]. Donc l'haplogroupe A réfère d'abord à un ensemble de lignées qui ne possèdent pas les marqueurs génétiques définissant l'haplogroupe BT (et ses dérivés), bien que beaucoup de lignées comprises dans l'haplogroupe A diffèrent par leur date d'origine.
Les mutations M91 et P97 distinguent l'haplogroupe A de l'haplogroupe BT. Dans les chromosomes haplogroupe A, le marqueur M91 consiste en une suite de 8 unités de base T, alors que dans l'haplogroupe BT et dans les chromosomes du chimpanzé, ce marqueur consiste en 9 unités de base T. Ceci suggère que la suite de 9T de l'haplogroup BT était la version ancestrale et que l'haplogroupe A fut formé par la délétion d'une base nucléique[10],[1].
Mais selon Fulvio Cruciani et al. 2011, la région entourant le marqueur M91 est un point chaud (hotspot) de mutations sujet aux mutations récurrentes. Il est donc possible que la suite de 8T de l'haplogroupe A soit l'état ancestral de M91 et 9T de l'haplogroupe BT soit l'état dérivé qui est apparu par une insertion de 1T. Ceci expliquerait pourquoi les subclades A1b et A1a-T, les branches les plus anciennes de l'haplogroupe A, possèdent toutes deux 8T. En outre Cruciani et al. 2011 ont déterminé que le marqueur P97, qui est aussi utilisé pour identifier l'haplogroupe A, posséderait l'état ancestral dans l'haplogroupe A mais l'état dérivé dans l'haplogroupe BT[1].
Les premières études rapportaient que les lignages de l'haplogroupe A avaient émergé il y a environ 60 000 ans, ce qui était bien plus récent que la date de plus récent ancêtre pour les lignages de l'ADN mitochondrial, dont la date de coalescence, l'ancienneté fournie par la statistique sur la diversité génétique, donne entre 150 000 et 200 000 ans, ce qui était quelque peu étrange. Mais Cruciani et al. 2011 ont repoussé la date de la racine de l'arbre du chromosome-Y il y a 142 000 ans[1].

Origine
Plusieurs propositions pour l'origine de l'haplogroupe A suggèrent qu'il était associé à la population ancestrale de chasseurs-cueilleurs d'Afrique australe, du fait que l'haplogroupe A est fréquent chez le peuple San. En plus, les lignages de l'ADN mitochondriale les plus anciens sont aussi principalement présents dans la population San. Il est plus probable d'avoir un parallélisme d'évolution des mutations dans les lignées paternelles et maternelles qu'une divergence.
Cependant les lignages A d'Afrique australe sont des subclades des lignages A basaux trouvés dans d'autres parties de l'Afrique. Ceci suggère que les lignages A sont arrivés en Afrique australe en provenance d'ailleurs[11]. Les deux lignages les plus anciens de l'haplogroupe A, A1b et A1a, ont été détectés en Afrique de l'Ouest, du Nord-Ouest et centrale. Fulvio Cruciani et al. ont suggéré que ces lignages pourraient avoir émergé quelque part entre l'Afrique centrale et le Maghreb, quoiqu'une telle interprétation en est encore au premier stade à cause de l'incomplète couverture géographique de l'Afrique des études du chromosome Y[1].
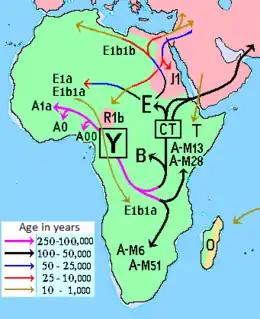
Distribution
Dans un échantillon composite de 3551 Africains hommes, l'haplogroupe A y avait une fréquence de 5,4 %[12]. Les plus hautes fréquences de l'haplogroupe A sont rapportées chez les Khoïsan d'Afrique australe, les Falashas, et les locuteurs de langues nilo-sahariennes des Soudan et Sud-Soudan.
Afrique centrale
| Afrique | ||
| Population | Fréq. (en %) | |
|---|---|---|
| [8] | San Tsumkwe (Namibie) | 66% |
| [8] | Nama (Namibie) | 64 |
| [13] | Dinka (Sud-Soudan) | 62 |
| [13] | Shilluk (Sud-Soudan) | 53 |
| [13] | Nouba (Soudan) | 46 |
| [14] | Khoïsan | 44 |
| [15],[16] | Falashas | 41 |
| [8],[15] | Kung/Sekele | ~40 |
| [13] | Maba (Tchad) | 35 |
| [13] | Nuer (Sud-Soudan) | 33 |
| [13] | Four (Soudan) | 31 |
| [8] | Maasai (Kenya) | 27 |
| [17] | Nara (Érythrée) | 20 |
| [13] | Masalit (Soudan) | 19 |
| [8],[18] | Amhara (Éthiopie) | ~16 |
| [14] | Éthiopiens | 14 |
| [19] | Bantou (Kenya) | 14 |
| [8] | Mandara (Cameroun) | 14 |
| [13] | Haoussa (Soudan) | 13 |
| [15] | Khwe (Afrique du Sud) | 12 |
| [15] | Foulbé (Cameroun) | 12 |
| [8] | Damara (Namibie) | 11 |
| [18] | Oromo (Éthiopie) | 10 |
| [17] | Kunama (Érythrée) | 10 |
| [8] | Sud Sémitique (Éthiopie) | 10 |
| [19] | Arabes (Égypte) | 3 |
L'haplogroupe A3b2-M13 est observé dans les populations du Nord-Cameroun (2/9 = 22 % Tupuri[8], 4/28 = 14 % Mandarawa[8], 2/17 = 12 % Foulbé[15]) et dans l'Est du Congo-Kinshasa (2/9 = 22 % pour les Alurs[8], 1/18 = 6 % les Hemas[8], 1/47 = 2 % les Pygmées Mbuti[8]).
L'haplogroupe A-M91(xA1a-M31, A2-M6/M14/P3/P4, A3-M32) (la mutation M91 présente avec aucune des mutations M31,M6,M14,P3,P4 et M32 présentes) est observé chez les Bakolas du Sud Cameroun (3/33 = 9 %)[8].
Sans avoir testé les subclades, l'haplogroupe A est observé par des prélèvements dans plusieurs populations du Gabon, avec 9 % (3/33) chez les Bakas, 3 % (1/36) Ndumus, 2 % (1/46) Adoumas, 2 % (1/57), Nzebis, et 2 % (1/60) Tshogos[6].
Afrique de l'Est
L'haplogroupe A3b2-M13 est commun chez les Sud-Soudanais (53 %)[13], spécialement chez les [Dinkas] (16/26 - 61,5 %)[13]. L'haplogroupe A3b2-M13 est aussi observé dans d'autres populations du Sud-Soudan à une fréquence de 45 % (18/40), dont 1/40 A3b2a-M171[14]. l'haplogroupe A a aussi été rapporté chez 14,6 % (7/48) des Amharas[18], 10,3 % (8/78) chez les Oromos[18], 13,6 % (12/88) dans une autre population d'Éthiopie[14], et 41 % chez les Falashas (Cruciani et al. 2002), et d'importants pourcentages sont aussi partagés chez les Bantous au Kenya (14 %, Luis et al. 2004) et les Iraqw en Tanzanie (3/43 = 7,0 % (Luis et al. 2004) à 1/6 = 17 % (Knight et al. 2003)).
Afrique du Nord
Le subclade A1 est observé chez les Berbères du Maroc, tandis que le subclade A3b2 est observé chez environ 3 % des Égyptiens hommes.
Afrique australe
Une étude a trouvé l'haplogroupe A chez les tribus parlant une langue khoïsan avec des fréquences allant de 10 % à 70 %[8]. De manière surprenante, l'haplogroupe n'a pas été trouvé chez les Hadzas de Tanzanie, une population traditionnellement considérée comme une survivance des Khoïsan à cause de la présence de clics consonants dans leur langue.
Eurasie
La forme A1 de l'haplogroupe A a été observée chez des hommes européens en Angleterre. Comme A3b2, on l'a observé avec une fréquence basse en Asie Mineure, au Moyen-Orient, et dans quelques iles méditerranéennes, chez les Turcs des côtes de la mer Égée, les Sardes, les Palestiniens, les Jordaniens, les Yéménites, et les Omanis. Sans avoir testé aucun subclade, l'haplogroupe A est observé chez les Grecs de Mytilène dans l'île égéenne de Lesbos[20] et au Sud et Centre Portugal et Madère[21]. Les auteurs d'une étude ont rapporté avoir trouvé ce qui apparaissait être l'haplogroupe A dans 3,1 % (2/65) des Chypriotes[22], sans toutefois exclure définitivement la possibilité que l'un ou l'autre de ces individus puisse appartenir aux haplogroupes B ou C.
Sous-clades
A00
| Arbre de la famille de l'haplogroupe A | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Cet arbre phylogénétique des subclades de l'haplogroupe A est basé sur l'arbre YCC 2008[10] et des recherches publiées s'y rapportant. |
On doit à Mendez et al. en 2013 la découverte d'un haplogroupe jusque-là inconnu, qu'ils ont dénommé A00[26]. Voici le tableau des estimations pour la date de son apparition :
| Intervalle de confiance à 95% | Moyenne | Référence | Année de Publication |
|---|---|---|---|
| 237 (min) - 581 (max) | 338 | Mendez et al.[26] | 2013 |
| 192 - 307 | 254 | Karmin et al.[27] | 2015 |
| 253 - 343 | 291 | Trombetta et al.[28] | 2015 |
| 213 - 293 | 249 | Petr et al.[29] | 2020 |
L'intervalle de confiance compatible à toutes les publications est de 293 à 253 ka (293000 à 253000 ans). On sait que les Homo sapiens archaïques étaient déjà présent vers 300 ka au Djebel Irhoud au Maroc, toutefois la région d'identification du A00 est celle de l'Afrique centrale de l'Ouest.
Cet haplogroupe jusque-là inconnu a été découvert en 2012, dans le chromosome Y d'un afroaméricain dénommé Perry qui avait soumis son ADN pour une analyse généalogique commerciale[30]. La découverte ultérieure d'autres membres de l'haplogroupe A00 a conduit au classement du chromosome Y de Perry comme A00a (A-L1149).
Des recherches ont montré qu'environ 10 % des hommes Mbo du Cameroun occidental appartenaient à un haplogroupe A00b (A-A4987).
D'autres recherches effectuées en 2015 indiquent que la population moderne présentant le taux d'A00 le plus élevé est celle des Bangwa, un groupe de camerounais parlant le yemba, avec un taux de l'ordre de 40 % d'A00a (A-L1149). Par ailleurs, un homme Bangwa ne correspondait ni à A00a ni à A00b[31].
A2-T- P108
En 2007, sept hommes du Yorkshire, en Angleterre partageant le patronyme furent identifiés comme du sous-groupe A2-T de l'haplogroupe A. On découvrit que ces hommes avaient un ancêtre commun par la lignée masculine au XVIIIe siècle, mais aucune autre information sur un ancêtre africain ne fut trouvée. Le sous-groupe A-P108 est extrêmement rare. En plus des sept hommes du Yorkshire, on a trouvé seulement 25 autres porteurs vivants dans le sous-groupe A-P108, tous d'ascendance ouest-africaine[12],[32].
A1b-V164
Trouvé seulement chez les Pygmées Bakola (Sud-Cameroun) pour 8,3 % et les Berbères d'Algérie pour 1,5 %[1].
A1a-M31
Le subclade A1a-M31 est trouvé dans approximativement 2,8 % (8/282) d'un ensemble de sept échantillons de divers groupes ethniques en Guinée-Bissau, spécialement parmi les Papel-Manjaco-Mancanha (5/64 = 7,8 %)[7]. Précédemment, dans une étude publiée en 2003, Gonçalves et al. rapportaient 5,1 % (14/276) pour A1a-M31 en Guinée-Bissau et 0,5 % (1/201) aux îles du Cap-Vert[33]. Les auteurs d'une autre étude ont rapporté 5 % (2/39) pour A1a-M31 chez les Mandingues de Sénégambie et 2 % (1/55) chez les Dogons du Mali[8]. L'Haplogroup A1a-M31 est aussi présent chez 3 % (2/64) de Berbères du Maroc[15] et 2,3 % (1/44) d'un échantillon d'affiliation ethnique non spécifiée au Mali[14].
A2-M6
Ce subclade de l'haplogroupe A est trouvé typiquement parmi les peuples Khoïsan. Les auteurs d'une étude rapportent l'haplogroupe A2-M6(xA2b-P28) (ceci veut dire qu'on y exclut son sous-groupe A2b-P28 donné à part) pour 28 % (8/29) chez les San Tsumkwe et pour 16 % (5/32) chez les Kung/Sekele, et A2b-P28 pour 17 % (5/29) des San Tsumkwe, 9 % (3/32) des Kung (peuple)/Sekele, 9 % (1/11) des Namas, et 6 % (1/18) des Damaras[8]. Dans une autre étude, les auteurs rapportent pour A2 dans 15,4 % (6/39) des hommes Khoïsan, incluant 5/39 A2-M6/M14/M23/M29/M49/M71/M135/M141(xA2a-M114) (donc les A2 portant toutes les 8 mutations citées mais sans les porteurs de M114 donnés à part) et 1/39 A2a-M114[14].
A3-M32
Ce clade contient les branches les plus populeuses de l'haplogroupe A et se trouve principalement en Afrique de l'Est et Afrique australe.
A3a-M28
Ce subclade de l'haplogroupe A3 est seulement et rarement observé dans la Corne de l'Afrique. Dans 5 % (1/20) d'un mélange de locuteurs des langues sud-sémitiques d'Éthiopie[8], 1,1 % (1/88) d'Éthiopiens[14], et 0,5 % (1/201) de Somalis[4].
A3b1-M51
Ce subclade del'haplogroupe A3 est rencontré plus fréquemment parmi les peuples du groupe Khoïsan (6/11 = 55 % Nama[8] 11/39 = 28 % Khoïsan[14], 7/32 = 22 % Kung/Sekele[8], 6/29 = 21 % San Tsumkwe[8], 1/18 = 6 % Damaras[8]). Cependant il est aussi trouvé à de plus basses fréquences parmi les Bantous d'Afrique du Sud, comprenant 2/28 = 7 % Sotho–Tswana[8], 3/53 = 6 % Africains du Sud non-Khoïsan[14], 4/80 = 5 % Xhosas[8], et 1/29 = 3 % Zoulous[8].
A3b2-M13
Le subclade de l'haplogroupe A3 qui est communément trouvé en Afrique de l'Est et dans le Nord Cameroun (A3b2-M13) est différent de ceux trouvés chez les Khoïsan et est relié seulement de manière éloigné à ceux-ci (Il n'est réellement qu'un des nombreux subclades à l'intérieur de l'haplogroupe A). Cette découverte suggère une ancienne divergence.
Au Soudan, l'haplogroupe A3b2-M13 est trouvé chez 28/53 = 52,8 % des Sud-Soudanais, 13/28 = 46,4 % des Noubas du Soudan central, 25/90 = 27,8 % des Soudanais de l'Ouest, 4/32 = 12,5 % des Haoussas du Soudan, et 5/216 = 2,3 % des Soudanais du Nord[34].
En Éthiopie, une étude a rapporté 14,6 % (7/48) pour A3b2-M13 chez les Amharas et 10,3 % (8/78) chez les Oromos[18]. Une autre étude rapporte 6,8 % (6/88) pour A3b2b-M118 et 5,7 % (5/88) pour A3b2*-M13(xA3b2a-M171, A3b2b-M118) chez les Éthiopiens, donnant un total de 12,5 % (11/88) A3b2-M13[14].
L'haplogroupe A3b2 a été aussi observé occasionnellement hors d'Afrique centrale et orientale, comme sur la côte de la mer Égée en Turquie (2/30 = 6,7 %[35]), Juifs yéménites (1/20 = 5 %[16]), Égypte (4/147 = 2,7 %[19], 3/92 = 3,3 %[8]), Arabes Palestiniens (2/143 = 1,4 %[36]), Sardaigne (1/77 = 1,3 %[37], 1/22 = 4,5 %[14]), la capitale de la Jordanie, Amman (1/101=1 %[38]), et Oman (1/121 = 0,8 %[19]).
Arbre phylogénétique
Cet arbre des sous-clades de l'haplogroupe A est basé sur l'arbre défini par le « Y-Chromosome Consortium » (YCC)[39], le « ISOGG Y-DNA Haplogroup Tree »[5], et les recherches publiées s'y rapportant.
« Adam » chromosomique
A00 (AF6/L1284)
- A00a (L1149, FGC25576, FGC26292, FGC26293, FGC27741)
- A00b (A4987/YP3666, A4981, A4982/YP2683, A4984/YP2995, A4985/YP3292, A4986, A4988/YP3731)
A0-T (L1085)
- A0 (CTS2809/L991) ex-A1b
- A1 (P305) ex-A1a-T, A0 et A1b
- A1a (M31)
- A1b (P108) ex-A2-T
- A1b1 (L419/PF712)
- A1b1a (L602, V50, V82, V198, V224)
- A1b1a1 (M14) ex A2
- A1b1a1a (M6)
- A1b1a1a1 (P28) ex-A1b1a1a1b et A2b
- A1b1a1a (M6)
- A1b1a1 (M14) ex A2
- A1b1b (M32) ex-A3
- A1b1b1 (M28) ex-A3a
- A1b1b2 (L427)
- A1b1b2a (M51/Page42) ex-A3b1
- A1b1b2a1 (P291)
- A1b1b2b (M13/PF1374) ex-A3b2
- A1b1b2b1 (M118)
- A1b1b2a (M51/Page42) ex-A3b1
- A1b1a (L602, V50, V82, V198, V224)
- BT (M91)
- A1b1 (L419/PF712)
Notes et références
Notes
- Les autres mutations sont : M49, M71, M135, M141, M196, M206, M212, MEH1, P3, P4, P5, P36.1, PK1, P247, and P248
Références
- (en) Fulvio Cruciani et al., « A Revised Root for the Human Y Chromosomal Phylogenetic Tree: The Origin of Patrilineal Diversity in Africa », The American Journal of Human Genetics, vol. 88, no 6, (DOI 10.1016/j.ajhg.2011.05.002, lire en ligne, consulté le )
- Karmin et al., « A recent bottleneck of Y chromosome diversity coincides with a global change in culture », Genome Research, vol. 25, no 4, , p. 459–66 (PMID 25770088, PMCID 4381518, DOI 10.1101/gr.186684.114)
- L. Mendez et al., « The Divergence of Neandertal and Modern Human Y Chromosomes », The American Journal of Human Genetics, vol. 98, no 4, , p. 728–34 (PMID 27058445, PMCID 4833433, DOI 10.1016/j.ajhg.2016.02.023)
- (en) Khaled K. Abu-Amero et al., « Saudi Arabian Y-Chromosome diversity and its relationship with nearby regions », BMC Genetics, vol. 10, no 59, (PMID 19772609, DOI 10.1186/1471-2156-10-59, lire en ligne)
- (en) « Y-DNA Haplogroup Tree », International Society of Genetic Genealogy, (consulté le )
- (en) Gemma Berniell-Lee, Francesc Calafell, Elena Bosch et al., « Genetic and Demographic Implications of the Bantu Expansion: Insights from Human Paternal Lineages », Molecular Biology and Evolution, vol. 26, no 7, , p. 1581–1589 (PMID 19369595, DOI 10.1093/molbev/msp069, lire en ligne [EPUB], consulté le )
- (en) Alexandra Rosa, Carolina Ornelas, Mark A Jobling et al., « Y-chromosomal diversity in the population of Guinea-Bissau: a multiethnic perspective », BMC Evolutionary Biology, vol. 2007, no 7, , p. 124 (PMID 17662131, PMCID 1976131, DOI 10.1186/1471-2148-7-124)
- (en) Elizabeth T Wood, Daryn A Stove, Christopher Ehret et al., « Contrasting patterns of Y chromosome and mtDNA variation in Africa: evidence for sex-biased demographic processes », European Journal of Human Genetics, no 13, , p. 867–876, cf. Appendix A: Y Chromosome Haplotype Frequencies
- (en) P.A. Underhill, G. Passarino, A.A. Lin et al., « The phylogeography of Y chromosome binary haplotypes and the origins of modern human populations », Annals of Human Genetics, vol. 65, no 1, , p. 43–62 (PMID 11415522, DOI 10.1046/j.1469-1809.2001.6510043.x)
- (en) TM Karafet, FL Mendez, MB Meilerman, PA Underhill, SL Zegura et MF Hammer, « New binary polymorphisms reshape and increase resolution of the human Y chromosomal haplogroup tree », Genome Research, vol. 18, no 5, , p. 830–8 (PMID 18385274, PMCID 2336805, DOI 10.1101/gr.7172008, lire en ligne)
- (en) Batini et al., « Signatures of the pre-agricultural peopling processes in sub-Saharan Africa as revealed by the phylogeography of early Y chromosome lineages », Molecular Biology and Evolution, (DOI 10.1093/molbev/msr089, lire en ligne, consulté le )
- (en) TE King, EJ Parkin, G Swinfield et al., « Africans in Yorkshire? The deepest-rooting clade of the Y phylogeny within an English genealogy », Eur. J. Hum. Genet., vol. 15, no 3, , p. 288–93 (PMID 17245408, PMCID 2590664, DOI 10.1038/sj.ejhg.5201771)
- 28/53 (Dinka, Nuer, et Shilluk), (en) HY Hassan, PA Underhill, LL Cavalli-Sforza et ME Ibrahim, « Y-chromosome variation among Sudanese: restricted gene flow, concordance with language, geography, and history », Am. J. Phys. Anthropol., vol. 137, no 3, , p. 316–23 (PMID 18618658, DOI 10.1002/ajpa.20876, lire en ligne)
- (en) PA Underhill, P Shen, Lin et al., « Y chromosome sequence variation and the history of human populations », Nat. Genet., vol. 26, no 3, , p. 358–61 (PMID 11062480, DOI 10.1038/81685)
- (en) Fulvio Cruciani, Piero Santolamazza, Peidong Shen et al., « A Back Migration from Asia to Sub-Saharan Africa Is Supported by High-Resolution Analysis of Human Y-Chromosome Haplotypes », American Journal of Human Genetics, vol. 70, nos 1197–1214,
- (en) Peidong Shen, Tal Lavi, Toomas Kivisild et al., « Reconstruction of Patrilineages and Matrilineages of Samaritans and Other Israeli Populations From Y-Chromosome and Mitochondrial DNA Sequence Variation », Human Mutation, no 24, , p. 248-260
- (en) Fulvio Cruciani, Beniamino Trombetta, Daniele Sellitto et al., « Human Y chromosome haplogroup R-V88: a paternal genetic record of early mid Holocene trans-Saharan connections and the spread of Chadic languages », European Journal of Human Genetics, , p. 1–8
- (en) O Semino, AS Santachiara-Benerecetti, F Falaschi, LL Cavalli-Sforza et PA Underhill, « Ethiopians and Khoisan share the deepest clades of the human Y-chromosome phylogeny », Am. J. Hum. Genet., vol. 70, no 1, , p. 265–8 (PMID 11719903, PMCID 384897, DOI 10.1086/338306, lire en ligne)
- (en) J. R. Luis, D. J. Rowold, M. Regueiro et al., « The Levant versus the Horn of Africa: Evidence for Bidirectional Corridors of Human Migrations », American Journal of Human Genetics, vol. 74, nos 532–544,
- (en) F. Di Giacomo, F. Luca, N. Anagnou et al., « Clinal patterns of human Y chromosomal diversity in continental Italy and Greece are dominated by drift and founder effects », Molecular Phylogenetics and Evolution, vol. 28, no 3, , p. 387–395 (PMID 12927125, DOI 10.1016/S1055-7903(03)00016-2)
- (en) Rita Gonçalves, Ana Freitas, Marta Branco et al., « Y-chromosome Lineages from Portugal, Madeira and Açores Record Elements of Sephardim and Berber Ancestry », Annals of Human Genetics, vol. 69, no Pt 4, , p. 443–454 (PMID 15996172, DOI 10.1111/j.1529-8817.2005.00161.x)
- (en) C. Capelli, N. Redhead, V. Romano et al., « Population Structure in the Mediterranean Basin: A Y Chromosome Perspective », Annals of Human Genetics,
- group="N">Les autres mutations sont : M190, M220, et P289
- group="N">Les autres mutations sont : P100, P291
- group="N">Les autres mutations sont : M63, M127, M202, M219, M305
- Fernando Mendez, Thomas Krahn, Bonnie Schrack, Astrid-Maria Krahn, Krishna Veeramah, August Woerner, Forka Leypey Mathew Fomine, Neil Bradman, Mark Thomas, Tatiana M. Karafet et Michael F. Hammer, « An African American paternal lineage adds an extremely ancient root to the human Y chromosome phylogenetic tree », American Journal of Human Genetics, vol. 92, no 3, , p. 454–9 (PMID 23453668, PMCID 3591855, DOI 10.1016/j.ajhg.2013.02.002, lire en ligne).
- M. Karmin, Saag, Vicente et Wilson Sayres, « A recent bottleneck of Y chromosome diversity coincides with a global change in culture », Genome Research, vol. 25, no 4, , p. 459–66 (PMID 25770088, PMCID 4381518, DOI 10.1101/gr.186684.114).
- B. Trombetta, D'Atanasio, Massaia, Myres, Scozzari, Cruciani et Novelletto, « Regional Differences in the Accumulation of SNPs on the Male-Specific Portion of the Human Y Chromosome Replicate Autosomal Patterns: Implications for Genetic Dating », PLOS ONE, vol. 10, no 7, , e0134646 (PMID 26226630, PMCID 4520482, DOI 10.1371/journal.pone.0134646, Bibcode 2015PLoSO..1034646T).
- Martin Petr, Mateja Hajdinjak, Qiaomei Fu, Elena Essel, Hélène Rougier, Isabelle Crevecoeur, Patrick Semal, Liubov V. Golovanova, Vladimir B. Doronichev, Carles Lalueza-Fox, Marco de la Rasilla, Antonio Rosas, Michael V. Shunkov, Maxim B. Kozlikin, Anatoli P. Derevianko, Benjamin Vernot, Matthias Meyer, Janet Kelso, « The evolutionary history of Neanderthal and Denisovan Y chromosomes », Science, volume 369, numéro 6511, pages 1653-1656, .
- Descendant d'Albert Perry, un esclave né aux États-Unis entre vers 1819-1827, vivant dans le comté de York, en Caroline du Sud. Voir FamilyTreeDNA, Haplogroup A chart
- Which of Cameroon's peoples have members of haplogroup A00? // experiment.com update of funded research (Schrack/Fomine Forka) available online Quotes: We can now clearly see that with 40% A00, the Bangwa represent the epicentre of A00 in this region, and very possibly in the world. As I shared in the last Lab Note, we found that so far there are two main subgroups of A00, defined by different Y-SNP mutations, which, naturally, divide along ethnic lines: A00a among the Bangwa, and A00b among the Mbo. We also found the one Bangwa sample which didn't belong to either subgroup.".
- Article de journal: (en) « Yorkshire clan linked to Africa », sur BBC News, (consulté le )
- Rita Gonçalves, Alexandra Rosa, Ana Freitas et al., « Y-chromosome lineages in Cabo Verde Islands witness the diverse geographic origin of its first male settlers », Human Genetics, no 113, , p. 467–472
- (en) HY Hassan, PA Underhill, LL Cavalli-Sforza et ME Ibrahim, « Y-chromosome variation among Sudanese: restricted gene flow, concordance with language, geography, and history », Am. J. Phys. Anthropol., vol. 137, no 3, , p. 316–23 (PMID 18618658, DOI 10.1002/ajpa.20876, lire en ligne). Le groupe « sud-Soudan » comprend 26 Dinkas, 15 Shilluks, et 12 Nuers. Le groupe des « Soudanais de l'Ouest » comprend 26 Borgus, 32 Masalits, et 32 Fours. Le groupe des « Soudanais du Nord » comprend 39 Nubiens, 42 Bejas, 33 Coptes, 50 Gaaliens, 28 Meserias, et 24 Arakiens.
- (en) Cengiz Cinnioglu, Roy King, Toomas Kivisild et al., « Excavating Y-chromosome haplotype strata in Anatolia », Human Genetics, vol. 114, , p. 127–148 (DOI 10.1007/s00439-003-1031-4)
- (en) Almut Nebel, Dvora Filon, Bernd Brinkmann et al., « The Y Chromosome Pool of Jews as Part of the Genetic Landscape of the Middle East », American Journal of Human Genetics, vol. 69, , p. 1095–1112
- (en) Ornella Semino, Giuseppe Passarino, Peter J. Oefner et al., « The Genetic Legacy of Paleolithic Homo sapiens sapiens in Extant Europeans: A Y Chromosome Perspective », Science, vol. 290,
- (en) C Flores, N Maca-Meyer, JM Larruga, VM Cabrera, N Karadsheh et AM Gonzalez, « Isolates in a corridor of migrations: a high-resolution analysis of Y-chromosome variation in Jordan », J. Hum. Genet., vol. 50, no 9, , p. 435–441 (PMID 16142507, DOI 10.1007/s10038-005-0274-4)
- (en) Thomas Krahn, « YCC Tree », Houston, Texas, FTDNA (consulté le )
Annexes
Articles connexes
|
Haplogroupes du chromosome Y (Y-ADN) | ||||||||||||||||||||||||
| Plus récent ancêtre patrilinéaire commun | ||||||||||||||||||||||||
| A | ||||||||||||||||||||||||
| BT | ||||||||||||||||||||||||
| B | CT | |||||||||||||||||||||||
| DE | CF | |||||||||||||||||||||||
| D | E | C | F | |||||||||||||||||||||
| G | H | IJK | ||||||||||||||||||||||
| IJ | K | |||||||||||||||||||||||
| I | J | LT | K2 | |||||||||||||||||||||
| I1 | L | T | MS | P | NO | |||||||||||||||||||
| M | S | Q | R | N | O | |||||||||||||||||||
| R1 | R2 | |||||||||||||||||||||||
| R1a | R1b | |||||||||||||||||||||||
- Généralités :
- Génétique du chromosome-Y :
- Table de conversion des haplogroupes chromosome-Y
Liens externes
- (en) African Haplogroup project sur le site FamilyTreeDNA
- (en) Haplogroup A sur le site du National Geographic
- Portail de la Préhistoire
- Portail de l’anthropologie
- Portail de la biologie