Lignée cellulaire (développement)
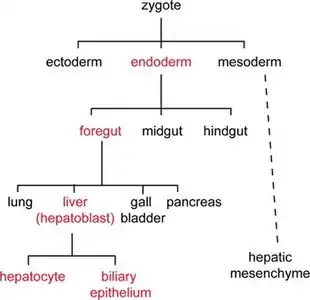
La lignée cellulaire est un concept qui définit l’historique d’une cellule par la succession de divisions mitotiques à partir de sa cellule souche embryonnaire jusqu’à l’organe final[1]. L’étude des lignées cellulaires d’un organisme mène à la formation de diagrammes, ou cartes de destin cellulaire (fate map), qui aident à comprendre les interactions entre les cellules, les déplacements cellulaires dans l’organisme ainsi que l’action des différents gènes développementaux dès la fertilisation de l’œuf. La comparaison entre les lignées cellulaires de différentes espèces permet d’éclaircir plusieurs mécanismes développementaux et évolutionnaires[2]. Le concept de base de cette discipline est qu’à partir de la fertilisation de l’œuf, une série de divisions mitotiques se produisent. Chez les animaux, en fonction de l’organisme, les différents feuillets embryonnaires (germ-layer), mésoderme, endoderme et ectoderme seront formés. Les cellules se différencient tôt ou tard en l'un de ces feuillets qui aura ensuite la capacité de former des tissus spécifiques. Chez les plantes, la segmentation du zygote peut être régulière et on parle de développement leptembryé, ou irrégulière et on parle de développement pachyembryé.
Historique
Laboratoire de biologie marine de Woods Hole
Les études sur la lignée cellulaire ont émané de l’embryologie et de l’étude des premiers clivages de l’œuf à la fin du XIXe et au début du XXe siècle. Le pionnier du domaine fut Charles Otis Whitman qui, par ses travaux sur Clepsine marginata puis C. complanata, C. bioculata et C. heteroclita, affirmait qu’un seul et même blastomère (cellule dérivant des premières divisions cellulaires d’un zygote) pouvait être à la base d’un feuillet embryonnaire entier comme l’ectoderme, l’endoderme ou le mésoderme puis à un organe complet bien précis[3]. Une génération de chercheurs œuvrant au Marine Biological Laboratory de Woods Hole lui a succédé, tels qu’Edmund B. Wilson, Edwin G. Conklin et bien d’autres, afin de comprendre et d’établir le concept de lignée cellulaire. E.B. Wilson, à l’origine, se concentrait sur les germ-layers et plus spécifiquement sur le mésoderme à travers ses travaux d’embryologie sur les annélides[4]. Il a démontré que la division cellulaire était déterminée par deux facteurs : une destinée préprogrammée et l’influence des cellules voisines[3]. De son côté, E. G. Conklin, par l’étude des lignées cellulaires des tunicates et gastéropodes, a mis en évidence le clivage déterminé et indéterminé[3],[5].
La teinture de Vogt
Les avancées majeures effectuées par ces chercheurs sont issues d'observations directes. À cette époque, le choix d'organismes et de phases de développement à étudier était limité. En effet, suivre une cellule à travers un organisme opaque n’était pas possible et une fois le développement avancé, le nombre de cellules en rendait le suivi très difficile[6]. En 1929, Walter Vogt développe une technique pour pallier ces problèmes. Vogt eut l'idée de teindre certaines cellules, permettant de suivre le mouvement des cellules teintées dans l’embryon. Il était ainsi en mesure de produire des cartes de destin cellulaire qui schématisent le devenir de chaque région d’un œuf. Cette stratégie avait cependant l'inconvénient de ne pas pouvoir suivre une cellule précise mais seulement un groupe de cellules, car la teinture était hydrosoluble et se dispersait dans les cellules voisines[6].
Thymidine tritiée
Plusieurs autres techniques ont été développées par la suite pour pallier ce problème, comme les teintures liposolubles ou les marqueurs nucléaires dont la thymidine tritiée (tritium-labeled thymidine). Hebert Taylor, en 1957, utilise pour la première fois ce traceur nucléaire radioactif, qui est en fait une molécule de thymidine marquée au tritium. La thymidine est l'un des quatre nucléotides qui composent l’ADN. Le tritium est un isotope de l’hydrogène, soit un atome d’hydrogène auquel on a ajouté deux neutrons. En exposant les cellules cibles à la thymidine tritiée, celles-ci intègrent la thymidine tritiée à leur ADN. On peut donc distinguer les cellules traitées des cellules non traitées, car ces premières seront radioactives. La thymidine radioactive marquée au carbone ou à l'azote avait déjà été utilisée mais n’était pas suffisante pour une visualisation microscopique autoradiographique. Le marquage au tritium permet une plus grande résolution et permet donc de suivre le déplacement et la division des cellules marquées plus efficacement[7]. Malgré la précision de cette technique, elle n'est efficace que sur une courte période de temps car le marqueur nucléaire est instable et se dilue à travers les multiples divisions cellulaires.
Chimère caille-poulet
En 1969, Nicole Le Douarin met au point une méthode utilisant des cellules chimères entre poulet et caille afin d’étudier le développement de la crête neurale du poulet. Cette technique tire avantage de la particularité du noyau des cellules de caille à l’interphase. Lors de cette phase, les cellules détiennent de l’hétérochromatine associée au nucléole en grande quantité, et possèdent une bonne affinité avec la teinture Feulgen-Rossenbeck. La chromatine des autres vertébrés ne se condense pas autant que celle de la caille à cette phase, ce qui permet d’en faire la distinction. Les cellules chimères teintes au Feulgen-Rossenbeck peuvent donc être distinguées des cellules non chimériques et deviennent alors un marqueur naturel. À l’époque de son développement, c’était la seule méthode de marqueur naturel pouvant être appliquée à des vertébrés supérieurs comme la souris et le poulet. La division et le déplacement des cellules de souris pouvaient maintenant être suivis grâce à leur caractéristique nouvellement acquise par la chimérisation avec les cellules de caille[8]. Cette façon de faire écarte le problème de la dilution des marqueurs nucléaires ou des teintures, mais reste encore limitée à des groupes de cellules et ne permet pas de suivre une lignée de cellules précises[6].
Caenorhabditis elegans
En 1977, la première carte de destin cellulaire d’un organisme entier de l’embryon à l’adulte a été complétée. Les travaux de Sulston et Horvitz ont permis d’achever ce projet en déterminant la lignée cellulaire post-embryonnaire de chacune des cellules de Caenorhabditis elegans. L’étude de la lignée cellulaire de C. elegans a été entamée par Sydney Brenner mais s’est limitée au stade embryonnaire. C’est lui qui a encouragé Sulston et Horvitz à étudier et terminer ce modèle. C. elegans est un modèle idéal pour ce genre d’étude car il est petit (environ 1 000 cellules non gonadiques), facile à cultiver (se développe de l’œuf à l’adulte en 3,5 jours), anatomiquement simple et génétiquement malléable. Les études ont été réalisées par observation directe, en utilisant un microscope à contraste interférentiel de Nomarski. En plaçant le nématode adéquatement sur un bloc d’agar sous ce microscope, il est possible de voir directement la migration, la division et la mort des différentes cellules. Cette technique a été utilisée ultérieurement par Sulston pour ses travaux en 1976, toujours sur C. elegans[9].
Injection de traceurs
Un peu plus tard, l’injection de traceurs directement dans la cellule cible a été développée. Cette technique permet enfin de se focaliser sur la lignée cellulaire d’une seule cellule, à condition d’y injecter le bon traceur. La peroxydase de raifort (horseradish peroxydase ou HRP) a d’abord été injectée pour étudier les stades embryonnaires précoces, mais s’est avérée insuffisante car cette molécule est sensible à la digestion protéolytique (digestion des protéines par des enzymes). David A. Weisblat, en 1980, a donc proposé la rhodamine-D-peptide (RDP) comme traceur fluorescent. Elle est formée par le couplage entre la rhodamine isothiocyanate (molécule fluorescente) et le dodécapeptide (Glu-Ala)2-Lys-Ala-(Glu-Ala)2-Lys-Gly. Ce dodécapeptide a été sélectionné par David A. Weisblat car il détient plusieurs caractéristiques favorables : une taille suffisante pour ne pas diffuser à travers les membranes ; un site de liaison pour la molécule fluorescente ; une réactivité aux fixateurs histologiques ; une résistance à la digestion protéolytique.
L’avantage de ce genre de traceurs est qu’ils peuvent être utilisés sur des organismes vivants, contrairement à l’HRP[10]. Cependant, cette technique n’est pas infaillible car elle souffre également de la dilution des molécules fluorescentes au fil des divisions cellulaires. De plus, injecter un traceur dans une cellule de petite taille peut être très complexe[6].
Vecteur rétroviral
Afin d’étudier les lignées cellulaires des stades plus avancés des vertébrés supérieurs, on a intégré l’utilisation de vecteurs rétroviraux pour implanter un marqueur dans une cellule plutôt que de l'y injecter directement. Le concept est assez simple : le vecteur doit intégrer le génome des cellules de façon que le gène du marqueur soit transmis génétiquement aux descendants. Plusieurs modifications du vecteur sont nécessaires pour le rendre fonctionnel. D’abord il doit contenir les gènes de base assurant sa transcription, son empaquetage et son intégration. Il doit aussi porter un gène marqueur comme le lacZ. Ce gène, lorsqu’exprimé, produit la β-galactosidase qui est facilement détectable car elle produit une coloration bleutée en présence de x-gal. C’est cette couleur qui permettra le suivi des cellules dans l’organisme. On doit ajouter un promoteur du lacZ au vecteur rétroviral car les promoteurs du rétrovirus sont supprimés. La capacité de réplication du rétrovirus doit être éliminée afin qu’il ne puisse pas se propager dans les cellules voisines. Autrement, il sera impossible de suivre une lignée cellulaire distincte[11].
Références
- (en) Robert C. King et Pamela K. Mulligan et William D. Stansfield, A Dictionary of Genetics, Oxford University Press, (DOI 10.1093/acref/9780199766444.001.0001, lire en ligne).
- (en) Hiroki Nishida et Thomas Stach, « Cell Lineages and Fate Maps in Tunicates: Conservation and Modification », Zoological Science, , p. 645-652 (DOI 10.2108/zs140117, lire en ligne)
- (en) Charles Galperin, « Cell Lineage to Developmental Genetics », History and Philosophy of the Life Sciences, , p. 301-344 (lire en ligne)
- (en) Edmund B. Wilson, The Embryology of the Earthworm, Journal of Morpholgy, , p. 387-462
- (en) Edwin G. Conklin, The Organization and Cell-Lineage of the Ascidian Egg, Philadelphie, , 119 p.
- (en) Claudio D. Stern et Scott E. Fraser, « Tracing the lineage of tracing cell lineages », Nature Cell Biology,
- (en) J. Herbert Taylor, Philip S. Woods et Walter L. Hughen, « The Organization and Duplication of Chromosomes as Revealed by Autoradiographic Studies Using Tritium-Labeled Thymidine », Biochemistry, , p. 122-128
- (en) Nicole Le Douarin, « Cell migration in early vertebrate develop- ment studied in interspecific chimeras », dans Katherine Elliott et Maeve O'Connor, Embryogenesis in Mammals, Elsevier, , 71-114 p. (lire en ligne).
- (en) J. E. Sulston et H. R. Horvitz, « Post-embryonic Cell Lineages of the Nematode, Caenorhabditis elegans », Developmental Biology, , p. 110-156
- (en) David A. Weisblat, Saul L. Zackson, Seth S. Blair et al., « Cell Lineage Analysis by Intracellular Injection of Fluorescent Tracers », Science, , p. 1538-1541
- (en) Joshua R. Sanes, John L. R. Rubenstein et Jean-François Nicolas, « Use of a recombinant retrovirus to study post-implantation cell lineage in mouse embryos », The EMBO Journal, , p. 3133-3142
Voir aussi
Bibliographie
- (en) Claudiu A. Giurumescu et Andrew D. Chisholm, « Cell Identification and Cell Lineage Analysis », Methods in Cell Biology, (lire en ligne).
- (en) Gunther S. Stent, « Developmental cell lineage », The International Journal of Developmental Biology, vol. 42, (lire en ligne [PDF], consulté le ).
- (en) Georgeta Crivat et Justin W. Taraska, « Imaging proteins inside cells with fluorescent tags », Trends in Biotechnology, (lire en ligne [PDF], consulté le ).
- (en) Gunther S. Stent, « The Role of Cell Lineage in Development », Philosophical Transactions of the Royal Society, vol. 312, no 1153 « Single Cell Marking and Cell Lineage in Animal Development », (lire en ligne, consulté le ).
- (en) Sally A. Moody, Cell Lineage and Fate Determination, Elsevier, (lire en ligne).
Liens externes
- (en) « Cell Lineage », sur Nature (consulté le ).
- Laboratoire Weisblat
- Lignée cellulaire de C. elegans
- Développement Animal
- Portail de la biologie cellulaire et moléculaire