Récepteurs olfactifs
Les récepteurs olfactifs sont des protéines transmembranaires localisés au pôle apical des neurones récepteurs olfactifs de l'épithélium olfactif ou de l'antenne selon l'espèce considérée. La détection d'indices chimiques dans l'environnement a un rôle essentiel pour la survie de la plupart des animaux en les informant sur la présence de nourriture, de partenaires reproductifs de prédateurs ou de pathogènes[1]. Les récepteurs olfactifs sont donc la porte d'entrée de signaux moléculaires en provenance de l'environnement ; ils permettent leur transduction en un message nerveux électrique interprétable par le système nerveux central. Les récepteurs olfactifs sont associés aux noms des Drs Linda B. Buck et Richard Axel, découvreurs en de la famille de gènes codant ces protéines[2] ; cette découverte leur valu le prix Nobel de physiologie ou médecine en .
Ces protéines présentent au niveau de leur extrémité N-terminale un domaine récepteur dirigé vers le milieu extracellulaire. Ce domaine est spécifique de certains composés chimiques. La multiplicité des récepteurs olfactifs - plusieurs centaines chez la majorité des vertébrés, appartenant à une super-famille de protéines couplées à des protéines G - et leur diversité permettent un encodage combinatoire des espèces moléculaires qui les atteignent. On compte ces dernières en dizaine de milliers, probablement toutes discriminées par le système olfactif de la plupart des vertébrés.
Le génome de la souris comporte environ 1 500 gènes codant des récepteurs olfactifs dont 80% sont transcrits[3],[4]. Chez l’homme, on compte environ 950 gènes dont 60% ne sont pas transcrits (pseudogènes)[4] et une centaine hérités de son ancêtre poisson, 350 environ étant fonctionnels, cette pseudogénisation pouvant être interprétée comme une réduction de l’univers olfactif humain ou une baisse des capacités de discrimination de son odorat[5]. Les récepteurs olfactifs forment la plus grande famille de gènes chez le mammifère, représentant entre 2% (comme chez l'Homme) et 5% du total des gènes codants selon les espèces[6].
Les cellules réceptrices olfactives se renouvellent tous les deux mois[7].
Évolution
La famille des gènes des récepteurs olfactifs chez les vertébrés a évolué à travers des événements génomiques tels que des duplications de gènes[8]. Des duplications en tandem ont été mises en évidence par le fait que de nombreux gènes codant des récepteurs appartenant à la même famille phylogénétique sont situés au même locus chromosomique[9]. Une telle évolution implique également l'élimination des gènes au sein des répertoires de famille multigénique par des mutations qui créent des pseudogènes non fonctionnels. Il a été proposé que l’évolution de la vision des couleurs chez les primates peut avoir diminué l'utilité du sens de l'olfaction par les primates, ce qui expliquerait le relâchement des pressions sélectives sur ces gènes et l'accumulation de pseudogènes récepteurs olfactifs chez les primates[10].
Il a été démontré que la sélection négative est toujours faible chez les humains modernes ce qui autorise la propagation de mutations délétères dans les populations actuelles. Il en résulte que le plateau de fonction minimale n'a pas encore été atteint chez l'Homme moderne, et donc que la capacité olfactive pourrait encore diminuer[11]. Ceci est considéré comme le premier indice scientifique de l'évolution future de la génétique humaine[11].
Mécanismes
Transduction du signal
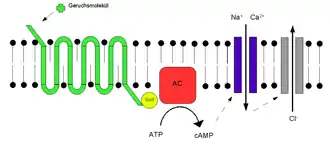
Les récepteurs olfactifs possèdent une affinité variable pour toute une gamme de molécules odorantes. Inversement, une seule molécule odorante peut se lier à plusieurs récepteurs olfactifs dotés d'affinités variables et qui dépendent des propriétés physico-chimiques de molécules, comme leurs volumes, leurs fonctions chimiques. Une fois que l'odorant s'est lié au récepteur olfactif, celui-ci subit des modifications structurelles. Il se lie et active une protéine G de type olfactif située à l'intérieur du neurone récepteur olfactif. La protéine G (Golf et/ou Gs) active à son tour la lyase - adénylate cyclase - qui convertit l'ATP en AMPc. L'AMPc entraîne l'ouverture de canaux ioniques laissant entrer les ions sodium () et calcium () dans la cellule conduisant à une dépolarisation du neurone. Cette dépolarisation est amplifiée par un courant chlorique () activé par le calcium. Cette dépolarisation, ou potentiel de récepteur, s'étend ensuite jusqu'au cône axonique du neurone récepteur olfactif où il y aura émission de potentiels d'action qui seront transmis au bulbe olfactif, premier relai de l'information olfactive au niveau du système nerveux central[12].
Le potentiel de récepteur revient au repos grâce à trois mécanismes. Des phosphodiestérases spécifiques participent à la dégradation de l'AMPc. Le forme un complexe avec la calmoduline qui va se lier au canal / et réduire son affinité pour l'AMPc (voir figure). Enfin, le est expulsé du neurone sous l'action d'un échangeur /[12].
Les séquences primaires de milliers de récepteurs olfactifs (RO) sont connues à partir des génomes de plus d'une douzaine d'organismes : ce sont des protéines transmembranaires à sept hélices, mais il n'y a (à compter de ) aucune structure connue d'aucun RO. Leurs séquences présentent des motifs GPCR de classe A typiques, utiles pour la construction de leurs structures par modélisation moléculaire. Golebiowski, Ma et Matsunami ont montré que le mécanisme de reconnaissance des ligands, bien que similaire à d'autres GPCR non olfactifs de classe A, implique des résidus spécifiques aux récepteurs olfactifs, notamment dans la sixième hélice. Il existe une séquence hautement conservée dans environ les trois quarts de toutes les salles d'opération qui est un site de liaison tripodal aux ions métalliques, et Suslick a proposé que les RO soient en fait des métalloprotéines (principalement avec des ions zinc, cuivre et éventuellement manganèse) qui servent de site acide de Lewis pour la liaison de nombreuses molécules odorantes[13]. Crabtree, en 1978, avait déjà suggéré que Cu (I) était "le candidat le plus probable pour un site métallo-récepteur dans l'olfaction" pour les substances volatiles à forte odeur qui sont également de bons ligands de coordination des métaux, tels que les thiols[14]. Zhuang, Matsunami et Block, en 2012, ont confirmé la proposition de Crabtree / Suslick concernant le cas spécifique d'une souris OU, MOR244-3, montrant que le cuivre est essentiel pour la détection de certains thiols et autres composés contenant du soufre[15]. Ainsi, en utilisant un produit chimique qui se lie au cuivre dans le nez de la souris, de sorte que le cuivre ne soit pas disponible pour les récepteurs, les auteurs ont montré que les souris ne pouvaient pas détecter les thiols. Cependant, ces auteurs ont également constaté que MOR244-3 n'avait pas le site de liaison spécifique aux ions métalliques suggéré par Suslick, mais qu'il présentait un motif différent dans le domaine EC2.
Le dysfonctionnement des métalloprotéines dans le système olfactif est supposé avoir un lien avec les maladies neurodégénératives à base amyloïde.
- Théorie vibratoire de l'olfaction (en)
Dans une étude controversée, il a également été proposé que les récepteurs olfactifs percevraient divers niveaux d’énergie vibratoire d’une molécule plutôt que des motifs structurels via des mécanismes de cohérence quantique[16]. Comme preuve, il a été mis en évidence que des drosophiles (mouches du vinaigre) pouvaient distinguer deux odorants ne différant que par la présence d'un isotope de l’hydrogène, le deutérium (qui modifiera radicalement les niveaux d’énergie vibratoire de la molécule)[17]. Non seulement les mouches pouvaient distinguer les formes deutériées et non deutériées d'un odorant, mais elles pouvaient également généraliser la propriété de "deutériosité" à d'autres molécules innovantes. En outre, ils ont généralisé le comportement d'évitement appris aux molécules non deutériées mais partageant un important étirement vibratoire avec les molécules deutérées, ce que la physique différentielle de la deutération (ci-dessous) a des difficultés à prendre en compte.
La deutération modifie les chaleurs d’adsorption et les points d’ébullition et de congélation des molécules (points d’ébullition: 100,0 °C pour H2O par rapport à 101,42 °C pour D2O; points de fusion: 0,0 °C pour H2O, 3,82 °C pour D2O), pKa ( par exemple, constante de dissociation : 9,71 × 10−15 H2O par rapport à 1,95 × 10−15 D2O, cf. l'eau lourde ) et la force de liaison hydrogène. De tels effets isotopiques sont extrêmement courants et il est donc bien connu que la substitution par le deutérium modifiera effectivement les constantes de liaison des molécules aux récepteurs protéiques.
Il a été affirmé que les récepteurs olfactifs humains sont capables de distinguer les isotopomères deutérés ou non de la cyclopentadécanone par détection de niveau d’énergie vibratoire[18]. Toutefois, cette affirmation a été contestée par un autre rapport selon lequel le récepteur reconnaissant le musc humain, OR5AN1, qui répond de manière robuste au cyclopentadécanone et à la muscone, ne permet pas de distinguer les isotopomères de ces composés in vitro[19]. En outre, le récepteur reconnaissant le (méthylthio) méthanethiol de souris, MOR244-3, ainsi que d’autres récepteurs olfactifs de souris et d’homme sélectionnés, ont réagi de manière similaire aux isotopomères normaux, deutérés et carbone 13 de leurs ligands respectifs, les résultats étant similaires à ceux trouvés avec le musk récepteur OR5AN1. Il a donc été conclu que la théorie des vibrations proposée ne s’appliquait pas au récepteur de musc humain OR5AN1, au récepteur de thiol de souris MOR244-3 ou à d’autres récepteurs olfactifs examinés. De plus, le mécanisme de transfert d'électrons proposé des fréquences vibratoires des substances odorantes pourrait être facilement supprimé par les effets quantiques des modes de vibration moléculaires non-odorants. De nombreux éléments de preuve vont donc à l’encontre de la théorie de l’odorat par vibration. Cette dernière étude a été critiquée car elle utilisait des "cellules dans un plat plutôt que dans des organismes entiers" et que "l'expression d'un récepteur olfactif dans des cellules rénales embryonnaires humaines ne reconstitue pas de manière adéquate la nature complexe de l'olfaction.... ". En réponse, les auteurs de la deuxième étude déclarent que" les cellules rénales embryonnaires ne sont pas identiques aux cellules du nez... mais si vous regardez les récepteurs, c'est le meilleur système du monde".
Notes et références
- (en) Kaupp UB., « Olfactory signalling in vertebrates and insects: differences and commonalities. », Nat Rev Neurosci., vol. 11, no 3, , p. 188-200 (PMID 20145624, DOI 10.1038/nrn2789, lire en ligne, consulté le )
- (en) Buck L et Axel R., « A novel multigene family may encode odorant receptors: a molecular basis for odor recognition. », Cell, vol. 65, no 1, , p. 175-87 (PMID 1840504, DOI 10.1016/0092-8674(91)90418-x., lire en ligne [PDF], consulté le )

- (en) Xinmin Zhang & Stuart Firestein, « The olfactory receptor gene superfamily of the mouse », Nature Neuroscience, no 5, , p. 124 - 133 (lire en ligne)
- Purves et al. 2005, p. 346-8.
- (en) G. Glusman, I. Yanai, I. Rubin et D. Lancet, « The Complete Human Olfactory Subgenome », Genome Research, vol. 11, no 5, , p. 685-702 (DOI 10.1101/gr.171001)
- (en) Kazushige Touhara, Atsushi Matsui et Yoshihito Niimura, « Extreme expansion of the olfactory receptor gene repertoire in African elephants and evolutionary dynamics of orthologous gene groups in 13 placental mammals », Genome Research, , gr.169532.113 (ISSN 1549-5469 et 1088-9051, PMID 25053675, DOI 10.1101/gr.169532.113, lire en ligne, consulté le )
- Lauralee Sherwood, Physiologie humaine, De Boeck Supérieur, (lire en ligne), p. 178
- (en) Nei M and Rooney AP, « Concerted and birth-and-death evolution of multigene families », Annu Rev Genet., vol. 39, , p. 121–152 (PMID 16285855, PMCID 1464479, DOI 10.1146/annurev.genet.39.073003.112240)
- (en) Niimura Y and Nei M, « Evolutionary dynamics of olfactory and other chemosensory receptor genes in vertebrates », J Hum Genet., vol. 51, no 6, , p. 505–517 (PMID 16607462, PMCID 1850483, DOI 10.1007/s10038-006-0391-8)
- (en) Gilad Y, Wiebe V, Przeworski M, Lancet D, Paabo S, « Loss of Olfactory Receptor Genes Coincides with the Acquisition of Full Trichromatic Vision in Primates », PLoS Biology., vol. 2, no 1, , p. 120–125 (PMID 14737185, PMCID 314465, DOI 10.1371/journal.pbio.0020005)
- (en) Pierron D, Cortés NG, Letellier T, Grossman LI., « Current relaxation of selection on the human genome: Tolerance of deleterious mutations on olfactory receptors. », Mol Phylogenet Evol.., vol. 66, no 2, , p. 558-564 (PMID 22906809, PMCID 314465)
- Purves et al. 2005, p. 345.
- (en) Wang J, Luthey-Schulten ZA et Suslick KS., « Is the olfactory receptor a metalloprotein? », Proc Natl Acad Sci U S A., vol. 100, no 6, , p. 3035-9 (PMID 12610211, PMCID 152240, DOI 10.1073/pnas.262792899, lire en ligne [PDF], consulté le )

- (en) Crabtree RH., « Copper (I): A possible olfactory binding site », J Inorg Nucl Chem., vol. 40, , p. 1453.
- (en) Duan X, Block E, Li Z, Connelly T, Zhang J, Huang Z, Su X, Pan Y, Wu L, Chi Q, Thomas S, Zhang S, Ma M, Matsunami H, Chen GQ et Zhuang H., « Crucial role of copper in detection of metal-coordinating odorants. », Proc Natl Acad Sci U S A., vol. 109, no 9, , p. 3492-7 (PMID 22328155, PMCID 3295281, DOI 10.1073/pnas.1111297109, lire en ligne [PDF])

- David Larousserie, « Quand les ondes montent au nez », Le Monde, (lire en ligne, consulté le ).
- (en) Franco MI, Turin L, Mershin A et Skoulakis EM., « Molecular vibration-sensing component in Drosophila melanogaster olfaction. », Proc Natl Acad Sci U S A, vol. 108, no 9, , p. 3797-802 (PMID 21321219, PMCID 3048096, DOI 10.1073/pnas.1012293108, lire en ligne [PDF], consulté le )

- (en) Gane S, Georganakis D, Maniati K, Vamvakias M, Ragoussis N, Skoulakis EM et Turin L., « Molecular vibration-sensing component in human olfaction. », PLoS One, vol. 8, no 1, , e55780 (PMID 23372854, PMCID 3555824, DOI 10.1371/journal.pone.0055780, lire en ligne [PDF], consulté le )

- (en) Block E, Jang S, Matsunami H, Sekharan S, Dethier B, Ertem MZ, Gundala S, Pan Y, Li S, Li Z, Lodge SN, Ozbil M, Jiang H, Penalba SF, Batista VS et Zhuang H., « Implausibility of the vibrational theory of olfaction. », Proc Natl Acad Sci U S A, vol. 112, no 21, , E2766-74 (PMID 25901328, PMCID 4450420, DOI 10.1073/pnas.1503054112, lire en ligne [PDF], consulté le )

Voir aussi
Bibliographie
- André Holley, « Système olfactif et neurobiologie », Terrain, no 47, , p. 107-122 (lire en ligne)
- Dale Purves, George J. Augustine, David Fitzpatrick, William C. Hall, Anthony-Samuel LaMantia, James O. McNamara et S. Mark Williams, Neurosciences, Bruxelles, De Boeck Université, coll. « Neurosciences & Cognition », , 3e éd., 811 p. (ISBN 978-2-8041-4797-6, lire en ligne), chap. 14 (« Les sens chimiques »), p. 346-8.
Articles connexes
- Dr Linda B. Buck
- Richard Axel
- Gène OR10C1
Liens externes
- Portail des neurosciences
- Portail de la biologie
- Portail de la biologie cellulaire et moléculaire
- Portail des odeurs, des senteurs et du parfum