Double hélice
En biologie moléculaire, la double hélice est la structure secondaire de l'ADN bicaténaire telle que l'a popularisée James Watson avec Francis Crick sur la base des travaux de cristallographie aux rayons X réalisés par Rosalind Franklin avec l'aide de Raymond Gosling, à travers son ouvrage La Double Hélice (en), publié en 1968.
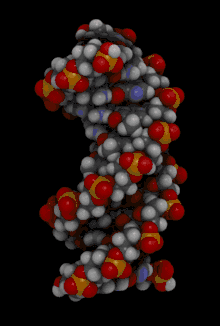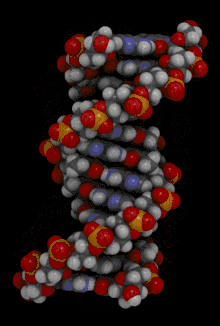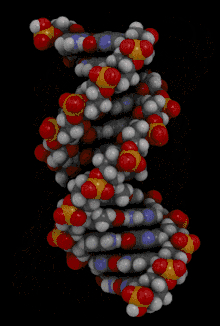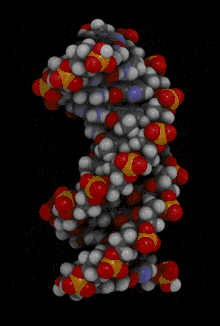
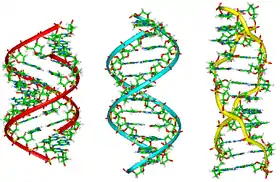
La double hélice d'ADN est souvent représentée à tort comme deux brins enroulés symétriquement l'un autour de l'autre ; en réalité, les trois principales formes d'ADN bicaténaire — dites ADN A, ADN B et ADN Z — présentent un grand et un petit sillon, ce qui conditionne largement leurs propriétés biochimiques : c'est en effet au niveau du grand sillon que les bases nucléiques sont le plus accessibles, notamment par les domaines de liaison à l'ADN de certaines protéines et enzymes.
| Paramètre | ADN A | ADN B | ADN Z |
|---|---|---|---|
| Sens de l'hélice | droite | droite | gauche |
| Motif répété | 1 bp | 1 bp | 2 bp |
| Rotation par paire de bases | 32,7° | 34,3° | 60°/2 |
| Paire de bases par tour d'hélice | 11 | 10,5 | 12 |
| Pas de l'hélice par tour | 2,82 nm | 3,32 nm | 4,56 nm |
| Allongement de l'axe par paire de bases | 0,24 nm | 0,32 nm | 0,38 nm |
| Diamètre | 2,3 nm | 2,0 nm | 1,8 nm |
| Inclinaison des paires de bases sur l'axe de l'hélice | +19° | −1,2° | −9° |
| Torsion moyenne (propeller twist) | +18° | +16° | 0° |
| Orientation des substituants des bases sur les résidus osidiques | anti | anti | Pyrimidine : anti, Purine : syn |
| Plissement / torsion endocyclique du furanose (Sugar pucker) | C3’-endo | C2’-endo | Cytosine : C2’-endo, Guanine : C2’-exo |
| Séquence | ΔG /kcal·mol−1 |
|---|---|
| T A | –0,19 |
| T G or C A | –0,55 |
| C G | –0,91 |
| A G or C T | –1,06 |
| A A or T T | –1,11 |
| A T | –1,34 |
| G A or T C | –1,43 |
| C C or G G | –1,44 |
| A C or G T | –1,81 |
| G C | –2,17 |
La stabilité de cette structure est déterminée par un ensemble de paramètres, notamment sa longueur, son taux de GC, sa séquence, sa concentration dans le solvant et la force ionique de celui-ci. En effet, dans la mesure où les deux brins d'ADN bicaténaire sont maintenus ensemble par les liaisons hydrogène entre paires de bases, plus il y a de paires de bases, plus il y a de liaisons hydrogène à briser. De plus, comme les paires adénine–thymine sont unies par deux liaisons hydrogène tandis que les paires guanine–cytosine sont unies par trois liaisons hydrogène, plus le taux de GC est élevé et plus la structure est stable. Enfin, la double hélice est également stabilisée par les interactions d'empilement entre bases nucléiques d'un même brin, de sorte que la séquence des brins influence également leur stabilité.
Notes et références
- (en) Richard R Sinden, DNA structure and function, Academic Press, , 398 p. (ISBN 0-12-645750-6)
- (en) Rich A, Norheim A, Wang AHJ, « The chemistry and biology of left-handed Z-DNA », Annual Review of Biochemistry, vol. 53, , p. 791–846 (PMID 6383204, DOI 10.1146/annurev.bi.53.070184.004043)
- (en) Ho PS, « The non-B-DNA structure of d(CA/TG)n does not differ from that of Z-DNA », Proc Natl Acad Sci USA, vol. 91, no 20, , p. 9549–9553 (PMID 7937803, PMCID 44850, DOI 10.1073/pnas.91.20.9549, Bibcode 1994PNAS...91.9549H)
- (en) Ekaterina Protozanova, Peter Yakovchuk et Maxim D. Frank-Kamenetskii, « Stacked–Unstacked Equilibrium at the Nick Site of DNA », Journal of Molecular Biology, vol. 342, no 3, , p. 775-785 (PMID 15342236, DOI 10.1016/j.jmb.2004.07.075, lire en ligne)
- Portail de la biologie cellulaire et moléculaire