Bactérie mangeuse de nylon
Les bactéries mangeuses de nylon forment une souche de Flavobacterium capable de digérer certains sous-produits de Nylon 6 (en). Cette souche de Flavobacterium sp. KI72, est devenue plus populaire sous le nom de « bactérie mangeuse de nylon », et les enzymes utilisées pour digérer des molécules de synthèse produites par l'humain sont devenues connues sous le nom de « nylonases[réf. nécessaire] ». De nombreux autres organismes présentent la même capacité[Lesquels ?].
Découverte
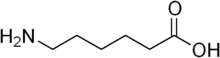
En 1975, une équipe de scientifiques japonais découvre une souche de Flavobacterium, vivant dans des bassins contenant des eaux usées à côté d'une usine de nylon, et capable de digérer certains sous-produits de nylon 6, tels que le dimère linéaire de 6-aminohexanoate. Ces substances ne sont pas connues pour avoir existé avant l'invention du nylon en 1935.
Une étude ultérieure[Laquelle ?] montre que chez ces bactéries, les trois enzymes utilisées pour digérer les produits dérivés sont significativement différentes de toutes les autres enzymes produites par d'autres souches de Flavobacterium (ou même de toutes les autres bactéries), et qu'elles ne sont pas efficaces sur les supports qui ne sont pas des sous-produits du nylon[1].
Recherches ultérieures
Cette découverte mène le généticien Susumu Ohno à publier un article en , dans lequel il suppose que le gène codant l'une des enzymes, l'acide 6-aminohexanoïque hydrolase, a pour origine la combinaison d'une duplication génétique avec une mutation du cadre de lecture[2]. Ohno suggère également que de nombreux nouveaux gènes ont évolué de cette façon.
Un article scientifique publié en 2007, qui décrit une série d'études menées au sein d'une équipe dirigée par Seiji Negoro de l'Université de Hyogo (Japon), suggère qu'en réalité, aucune mutation du cadre de lecture n'a été impliquée dans l'évolution de l'acide 6-aminohexanoïque hydrolase[3]. Cependant, de nombreux autres gènes ont par la suite été découverts, ayant évolué par duplication génétique suivie d'une mutation affectant au moins une partie du gène[citation nécessaire].
Un article de 1995 montre que les scientifiques ont également été en mesure de modifier une autre espèce de bactérie, Pseudomonas aeruginosa, afin de faire évoluer la capacité de dégrader les mêmes sous-produits du nylon en laboratoire, en les forçant à vivre dans un environnement ne comportant aucune autre source de nutriments. La souche P. aeruginosa ne semble cependant pas utiliser les mêmes enzymes que celles employées par la souche Flavobacterium[4].
D'autres scientifiques sont parvenus à produire des enzymes afin de les transférer de la souche de Flavobacterium à une souche de bactérie Escherichia coli par l'intermédiaire d'un plasmide de transfert[5].
De nombreux autres organismes se sont révélés capables de dégrader le Nylon 6 (en) : la bactérie Anoxybacillus rupiensis, mais aussi des Pseudomonas, Streptococcus, Staphylococcus, Micrococcus, Moraxella, certaines espèces d'Actinomycétes, le genre Saccharomonospora ainsi que des fungi (Aspergillus niger, Aspergillus glaucus),…[6]. Le fungi Geobacillus thermocatenulatus peut aussi dégrader le Nylon 12 (en) et le Nylon 66 (en)[6].
Rôle dans l'enseignement de l'évolution
Il existe un consensus scientifique pour dire que la capacité de cette bactérie à synthétiser de la nylonase est vraisemblablement dû à une mutation. Celle-ci a persisté chez cette souche grâce à l'amélioration de la valeur sélective conférée à la bactérie porteuse. L'enzyme permettant de dégrader le nylon a ainsi été produite par une mutation qui a totalement randomisé le gène original. Le nouveau gène a alors acquis une nouvelle capacité catalytique, bien qu'elle soit faible.
La bactérie mangeuse de nylon est considérée à la fois comme un bon exemple pour illustrer le fait que les mutations peuvent facilement constituer le support de l'évolution par la sélection naturelle, tout comme un exemple d'évolution récent, puisque le nylon, synthétique, n'a été créé que dans les années 1930[7],[8],[9],[10].
Notes et références
- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « Nylon-eating bacteria » (voir la liste des auteurs).
- Kinoshita, S., Kageyama, S., Iba, K., Yamada, Y., Kageyama, S., Kageyama, S., Kageyama, S., Kageyama, S., Kageyama, S. et Kageyama, S., « Utilization of a cyclic dimer and linear oligomers of e-aminocaproic acid by Achromobacter guttatus », Agricultural & Biological Chemistry, vol. 39, no 6, , p. 1219–23 (ISSN 0002-1369, DOI 10.1271/bbb1961.39.1219).
- Ohno S, « Birth of a unique enzyme from an alternative reading frame of the preexisted, internally repetitious coding sequence », Proc Natl Acad Sci USA., vol. 81, no 8, , p. 2421–5 (PMID 6585807, PMCID 345072, DOI 10.1073/pnas.81.8.2421, lire en ligne).
- « Nylon-oligomer degrading enzyme/substrate complex: catalytic mechanism of 6-aminohexanoate-dimer hydrolase », J. Mol. Biol., vol. 370, no 1, , p. 142–56 (PMID 17512009, DOI 10.1016/j.jmb.2007.04.043).
- « Emergence of nylon oligomer degradation enzymes in Pseudomonas aeruginosa PAO through experimental evolution », Appl. Environ. Microbiol., vol. 61, no 5, , p. 2020–2 (PMID 7646041, PMCID 167468, lire en ligne).
- « Plasmid-determined enzymatic degradation of nylon oligomers », J. Bacteriol., vol. 155, no 1, , p. 22–31 (PMID 6305910, PMCID 217646, lire en ligne).
- Mayaada S. Mahdi, Rasha S. Ameen et Hiba K. Ibrahim, « Study on Degradation of Nylon 6 by thermophilic bacteria Anoxybacillus rupiensis Ir3 (JQ912241) », International Journal of Advanced Research in Biological Sciences, vol. 3, no 9, (DOI 10.22192/ijarbs)
- Thwaites WM, « New Proteins Without God's Help », Creation Evolution Journal, National Center for Science Education (NCSE), vol. 5, no 2, , p. 1–3 (lire en ligne).
- Evolution and Information: The Nylon Bug.
- Why scientists dismiss 'intelligent design', Ker Than, MSNBC, Sept. 23, 2005.
- Miller, Kenneth R. Only a Theory: Evolution and the Battle for America's Soul (2008) pp. 80-82.
Annexes
Bibliographie
- (en) Shinichi Kinoshita, Sadao Kageyama, Kazuhiko Iba et Yasuhiro Yamada, « Utilization of a Cyclic Dimer and Linear Oligomers of ε-Aminocaproic Acid by Achromobacter guttatus KI 72 », Agricultural and Biological Chemistry, vol. 39, , p. 1219–1223 (DOI 10.1271/bbb1961.39.1219, lire en ligne, consulté le )
- (en) « No stop codons in the antisense strands of the genes for nylon oligomer degradation », Proc Natl Acad Sci USA., vol. 89, no 9, , p. 3780–4 (PMID 1570296, PMCID 525574, DOI 10.1073/pnas.89.9.3780, lire en ligne)
- (en) « Emergence of nylon oligomer degradation enzymes in Pseudomonas aeruginosa PAO through experimental evolution », Appl. Environ. Microbiol., vol. 61, no 5, , p. 2020–2 (PMID 7646041, PMCID 167468, lire en ligne)
Liens internes
- Portail de la microbiologie
- Portail origine et évolution du vivant