Néofonctionnalisation
La néofonctionnalisation est une des issues possibles de la divergence fonctionnelle, et a lieu lorsqu'une copie d'un gène, ou paralogue, prend en charge une fonction totalement nouvelle à la suite d'une duplication génique. La néofonctionnalisation est un processus de mutation adaptatif, ce qui signifie qu'une des copies du gène ancestral doit muter afin de développer une fonction qui n'existait pas chez ce gène ancestral[1],[2],[3]. En d'autres mots, l'un des duplicons conserve sa fonction originale, alors que l'autre accumule des modifications moléculaires qui font qu'avec le temps, ce dernier peut réaliser une fonction différente[4]. On pense que ce processus n'est pas affecté par la pression de sélection car une des copies du gène peut muter sans affecter de façon préjudiciable la fitness d'un organisme car la fonction ancestrale est conservée dans l'autre copie[5],[6],[7],[8].

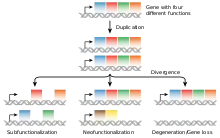
Processus
Le processus de la néofonctionnalisation part d'une duplication génique, et on pense qu'il a lieu en tant que mécanisme de défense contre l'accumulation de mutations délétères[6],[8],[9]. À la suite de cette duplication, il y a deux copies identiques du gène ancestral réalisant exactement la même fonction. Cette redondance permet à l'une des copies de prendre en charge une nouvelle fonction. Si cette dernière s'avère avantageuse, la sélection naturelle la sélectionne de façon positive et cette nouvelle mutation devient fixée dans la population[3],[10]. L'occurrence de la néofonctionnalisation peut le plus souvent être attribuée aux changements survenus dans la région codante ou dans les éléments régulateurs d'un gène[8]. Il est beaucoup plus rare d'observer des changements majeurs dans la fonction des protéines, comme au sein de la structure des sous-unités ou dans l'affinité entre un substrat et son ligand, comme résultat de la néofonctionnalisation[8].
Contraintes sélectives
La néofonctionnalisation est également communément appelée "mutation au cours de la non-fonctionnalité" ou "mutation au cours de la redondance"[11]. En dépit du cas où une mutation survient à la suite d'une non-fonctionnalité d'un gène ou est due à des copies géniques redondantes, l'important est que dans les deux scénarios, l'une des copies du gène est libérée des contraintes sélectives et acquiert au gré du hasard une nouvelle fonction, qui est ensuite améliorée par la sélection naturelle. On pense que ce processus a lieu très rarement au cours de l'évolution pour deux raisons majeures. La première est que les changements fonctionnels requièrent typiquement un grand nombre de changements d'acides aminés, ce qui a une faible probabilité d'arriver. La deuxième raison est que des mutations délétères surviennent plus fréquemment que des mutations avantageuses au cours de l'évolution. Cela rend beaucoup plus probable le fait que la fonction du gène soit perdue au cours du temps (pseudogénisation) que le fait qu'une nouvelle fonction génique voit le jour. Walsh a découvert que la probabilité relative de la néofonctionnalisation est déterminée par l'avantage sélectif et le rythme relatif des mutations avantageuses[12]. Cela a été prouvé par la dérivation de la probabilité relative de la néofonctionnalisation à la pseudogénisation, donnée par l'équation , où ρ est le rapport entre le taux de mutations avantageuses et le taux de mutations neutres, et S est la sélection de population, calculée comme 4 Ne S (Ne = taille efficace de la population, et S = intensité de la sélection)[12].
Modèle classique
En 1936, Muller a été le premier à proposer la néofonctionnalisation comme issue possible d'une duplication génique[13]. En 1970, Ohno a suggéré que la néofonctionnalisation était le seul mécanisme évolutif ayant donné naissance à de nouvelles fonctions géniques dans une population[8]. Il a également émis l'hypothèse que la néofonctionnalisation était la seule alternative à la pseudogénisation[2]. En 1987, Ohta était la première personne à suggérer que d'autres mécanismes puissent exister pour la préservation de gènes dupliqués dans la population[8]. Aujourd'hui, la sous-fonctionnalisation constitue une alternative à la néofonctionnalisation comme processus de fixation de duplicons qui fait largement consensus, et est actuellement la seule autre issue possible de la divergence fonctionnelle[2].
Néosubfonctionnalisation
La néosubfonctionnalisation a lieu lorsque la néofonctionnalisation constitue le résultat final d'une subfonctionnalisation. En d'autres mots, lorsqu'une duplication génique a lieu et forme des paralogues qui subissent une sous-fonctionnalisation, une des copies géniques continue son "voyage évolutif" et accumule des mutations donnant naissance à une nouvelle fonction[8],[14]. Certains chercheurs pensent que la néofonctionnalisation constitue l'étape finale de tous les gènes ayant subi une sous-fonctionnalisation. Par exemple, selon Rastogi et Liberles, "la néofonctionnalisation constitue le sort final de toutes les copies du gène dupliqué conservées dans le génome, et la sous-fonctionnalisation existe seulement en tant qu'état de transition afin de préserver la copie du gène dupliqué"[2]. Les résultats de leur étude sont ponctués à mesure que la taille de la population augmente.
Exemple de la protéine antigel
L'évolution de la protéine antigel chez les poissons zoarcidés de l'Antarctique fournit un bon exemple de néofonctionnalisation à la suite d'une duplication génique. Dans le cas de la protéine antigel de type III des poissons zoarcidés, le gène a divergé depuis une copie paralogue du gène codant l'acide sialique synthase (SAS)[15]. On a trouvé que le gène ancestral de la SAS possédait à la fois la fonction acide sialique synthase et une fonction rudimentaire de liaison à la glace polaire. Après duplication, l'un des paralogues a commencé à accumuler des mutations ayant mené au remplacement des domaines SAS du gène, permettant ensuite un développement et une optimisation de la fonction antigel[15]. Le nouveau gène est désormais capable de dépression non congressive du point de congélation, et est ainsi néofonctionnalisé[15]. Cette spécialisation permet à ces poissons de survivre aux températures glaciales des eaux de l'Antarctique.
Limites du modèle
Des limites existent dans la néofonctionnalisation en tant que modèle pour la divergence fonctionnale, principalement car :
- la quantité de changements nucléotidiques donnant naissance à une nouvelle fonction doit être la plus minime possible, rendant une pseudogénisation beaucoup plus probable que la néofonctionnalisation à la suite d'une duplication génique[8] ;
- à la suite d'une duplication génique, les deux copies peuvent subir une pression de sélection équivalente à celle que subit le gène ancestral, ce qui signifie qu'aucune des deux copies n'est nécessaire pour la néofonctionnalisation[8] ;
- dans de nombreux cas, une sélection Darwinienne positive présente une explication plus parcimonieuse pour la divergence de familles multigéniques[8].
Notes et références
- Dirk A. Kleinjan, Ruth M. Bancewicz, Philippe Gautier et Ralf Dahm, « Subfunctionalization of duplicated zebrafish pax6 genes by cis-regulatory divergence », PLoS genetics, vol. 4, no 2, , e29 (ISSN 1553-7404, PMID 18282108, PMCID PMC2242813, DOI 10.1371/journal.pgen.0040029, lire en ligne, consulté le )
- Shruti Rastogi et David A. Liberles, « Subfunctionalization of duplicated genes as a transition state to neofunctionalization », BMC Evolutionary Biology, vol. 5, , p. 28 (ISSN 1471-2148, DOI 10.1186/1471-2148-5-28, lire en ligne, consulté le )
- Bernard Conrad et Stylianos E. Antonarakis, « Gene duplication: a drive for phenotypic diversity and cause of human disease », Annual Review of Genomics and Human Genetics, vol. 8, , p. 17–35 (ISSN 1527-8204, PMID 17386002, DOI 10.1146/annurev.genom.8.021307.110233, lire en ligne, consulté le )
- (en) Susumu Ohno, Evolution by Gene Duplication, New York, Heidelberg, Berlin, Springer-Verlag, , p. 59-87
- (en) Marie Sémon et Kenneth H. Wolfe, « Preferential subfunctionalization of slow-evolving genes after allopolyploidization in Xenopus laevis », Proceedings of the National Academy of Sciences, vol. 105, no 24, , p. 8333–8338 (ISSN 0027-8424 et 1091-6490, PMID 18541921, DOI 10.1073/pnas.0708705105, lire en ligne, consulté le )
- Riet De Smet et Yves Van de Peer, « Redundancy and rewiring of genetic networks following genome-wide duplication events », Current Opinion in Plant Biology, genome studies molecular genetics, vol. 15, no 2, , p. 168–176 (DOI 10.1016/j.pbi.2012.01.003, lire en ligne, consulté le )
- J. Graham Ruby, Alexander Stark, Wendy K. Johnston et Manolis Kellis, « Evolution, biogenesis, expression, and target predictions of a substantially expanded set of Drosophila microRNAs », Genome Research, vol. 17, no 12, , p. 1850–1864 (ISSN 1088-9051, PMID 17989254, PMCID PMC2099593, DOI 10.1101/gr.6597907, lire en ligne, consulté le )
- (en) Dan Graur et Wen-Hsiung Li, Fundamentals of Molecular Evolution, Second Edition, Publishers, Sunderland, Massachusetts, Sinauer Associates, Inc.,
- (en) Grigoris D. Amoutzias, Ying He, Jonathan Gordon et Dimitris Mossialos, « Posttranslational regulation impacts the fate of duplicated genes », Proceedings of the National Academy of Sciences, vol. 107, no 7, , p. 2967–2971 (ISSN 0027-8424 et 1091-6490, PMID 20080574, DOI 10.1073/pnas.0911603107, lire en ligne, consulté le )
- (en) Hideki Innan, « Population genetic models of duplicated genes », Genetica, vol. 137, no 1, , p. 19 (ISSN 0016-6707 et 1573-6857, DOI 10.1007/s10709-009-9355-1, lire en ligne, consulté le )
- (en) A. Hughes, Adaptive Evolution of Genes and Genomes, New York, Oxford University Press,
- (en) Michael Lynch et Allan Force, « The Probability of Duplicate Gene Preservation by Subfunctionalization », Genetics, vol. 154, no 1, , p. 459–473 (ISSN 0016-6731 et 1943-2631, PMID 10629003, lire en ligne, consulté le )
- (en) H. J. Muller, « Bar Duplication », Science, vol. 83, no 2161, , p. 528–530 (ISSN 0036-8075 et 1095-9203, PMID 17806465, DOI 10.1126/science.83.2161.528-a, lire en ligne, consulté le )
- (en) Xionglei He et Jianzhi Zhang, « Rapid Subfunctionalization Accompanied by Prolonged and Substantial Neofunctionalization in Duplicate Gene Evolution », Genetics, vol. 169, no 2, , p. 1157–1164 (ISSN 0016-6731 et 1943-2631, PMID 15654095, DOI 10.1534/genetics.104.037051, lire en ligne, consulté le )
- (en) Cheng Deng, C.-H. Christina Cheng, Hua Ye et Ximiao He, « Evolution of an antifreeze protein by neofunctionalization under escape from adaptive conflict », Proceedings of the National Academy of Sciences, vol. 107, no 50, , p. 21593–21598 (ISSN 0027-8424 et 1091-6490, PMID 21115821, DOI 10.1073/pnas.1007883107, lire en ligne, consulté le )
- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « Neofunctionalization » (voir la liste des auteurs).
Voir aussi
Articles connexes
- Portail origine et évolution du vivant
- Portail de la biologie