Lactobacille
Lactobacillus
| Règne | Bacteria |
|---|---|
| Division | Firmicutes |
| Classe | Bacilli |
| Ordre | Lactobacillales |
| Famille | Lactobacillaceae |
Le Lactobacille (Lactobacillus) est un genre de bactéries à gram positif, de la famille des Lactobacillaceae.
Les lactobacilles sont des bactéries lactiques[1] :
- gram-positives ;
- immobiles, non flagellés, non sporogènes ;
- soit homofermentaires, produisant à partir du glucose plus de 85 % d'acide lactique, soit hétérofermentaires et produisant du CO2, de l'acide lactique, de l'éthanol (et/ou de l'acide acétique) en quantités équimolaires ;
- aérotolérantes ou anaérobies ;
- catalase (certains ont une pseudocatalase) ;
- ayant des besoins nutritionnels complexes (milieu riche en glucides, acides aminés, peptides, lipides, sels, vitamines).
Habitats
Les lactobacilles sont présents dans les milieux riches contenant des substrats glucidiques tels que les muqueuses intestinales, orales et vaginales des humains et des animaux, sur les plantes, les aliments d'origine végétale, les produits fermentés ou en décomposition, les eaux usées[1].
Ils contaminent fréquemment les produits alimentaires et sont agents d'aigrissement[2]. Mais ils ne sont jamais pathogènes.
Place des lactobacilles dans le microbiote intestinal
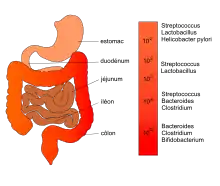
Tous les organismes supérieurs, mammifères, insectes et poissons hébergent des communautés bactériennes importantes. L'ensemble des microorganismes peuplant un organisme constitue son microbiote. Le principal microbiote humain se répartit dans la cavité orale et le long du tractus digestif mais c'est dans la partie distale du tube digestif que les bactéries sont en plus grand nombre. Elles sont disposées suivant un gradient oro-anal croissant avec une richesse microbienne maximale dans le côlon distal. Il y aurait en tout 1014 bactéries dans le tractus digestif[3] soit 10 à 20 fois le nombre total de cellules de tous les tissus du corps. Globalement, cette collectivité microbienne a une activité métabolique équivalente à un organe virtuel à l'intérieur d'un organe. L'humain héberge aussi d'autres communautés microbiennes importantes comme le microbiote cutané et le microbiote vaginal.
L'estomac, milieu fortement acide (jusqu'à pH 2) où l'oxygène est présent, héberge des microorganismes acido-tolérants et anaérobies facultatifs comme les streptocoques et les lactobacilles ainsi qu'un microaérophile, Helicobacter pylori[4]. Mais ils restent peu nombreux (101 à 103 cfu/ml) en raison de l'acidité et de l'activité motrice intestinale qui limite une colonisation stable de l'épithélium gastrique. Dans l'intestin grêle, le microbiote est constitué de bactéries anaérobies facultatives tels que les streptocoques, lactobacilles et entérobactéries et d'anaérobies strictes comme les bifidobactéries, les bactéroïdes et les clostridies. La flore reste encore relativement pauvre en raison du péristaltisme et de l'abondance des sécrétions ; la densité passe de 103 à 108 cfu/ml du duodénum à l'iléon. Dans le côlon[5], la flore microbienne est de plus en plus variée et abondante (1012 cfu/ml). Elle est dominée par les bactéries anaérobies strictes (Bacteroides 1011 par gramme de selle, Bifidobacterium, Clostridium) tandis que les bactéries anaérobies facultatives (comme les lactobacilles) disparaissent presque totalement.
Avant la mise au point de techniques de cultures anaérobies, les lactobacilles avaient la réputation d'être les organismes dominants de la flore intestinale, de l'estomac au jéjunum. Mais après 1960, la majorité des études moléculaires ont montré que les séquences de Lactobacillus ne représentaient en fait qu'une très petite proportion (moins de 1 %) de la population bactérienne. Il doit donc être considéré que la population de Lactobacillus qui peut être cultivée à partir de l'estomac et de l'intestin grêle est généralement très faible (< 104 bactéries par ml) et que la majorité de ces bactéries présentes transitent probablement à partir de la cavité orale et de la nourriture (Walter[6], 2008). L'analyse par le système PCR-DGGE des matières fécales[7] détecte la présence de bactéries lactiques (ici non cultivables) habituellement associées à la nourriture et aux levains utilisés dans l'alimentation (Lactobacillus Sakei (en)), Lb. curvatus). Il a été trouvé chez les individus ayant une population importante et stable de lactobacilles fécaux que ces individus pouvaient garder des souches particulières durant tout le long des 15 mois de l'étude. Il s'agissait de Lb. ruminis et de Lb. salivarius.
L'écosystème microbien intestinal, quoique dynamique, reste relativement stable. Les grands groupes de bactéries tels que les bifidobactéries, les bactéroïdes et les clostridies font preuve d'une grande stabilité au cours du temps jusqu'au niveau des espèces. Par contre, la population de lactobacilles dans les échantillons fécaux est fluctuante. Beaucoup de sujets traversent des périodes durant lesquelles aucun lactobacille n'est détectable[6].
De nombreuses espèces de lactobacilles ont été détectées dans la salive, les principales étant[8] Lactobacillus fermentum, Lb. rhamnosus, Lb. salivarius, Lb. casei et Lb. acidophilus. Une corrélation forte a été établie entre la quantité de Lactobacillus dans la salive et les caries dentaires.
En outre, le nombre de lactobacilles de la salive quoique variable, peut atteindre 105 cgu/ml. Sachant que cette salive produite à raison de 1 à 1,5 litre par jour est avalée, on comprend qu'elle puisse introduire dans le tractus digestif des doses de lactobacilles oraux significatives. Sur les 17 espèces de Lactobacillus associées avec le tractus digestif, une bonne partie ne sont donc pas autochtones[N 1] mais transitent à partir de l'alimentation ou de la salive.
Utilisations dans la production d'aliments fermentés
Quelques espèces de lactobacilles interviennent pour la production de produits alimentaires fermentés comme le yaourt, le fromage, le vin, la choucroute, les pickles, ainsi que pour l'ensilage. Le pain au levain est fabriqué en utilisant une « culture de départ » qui est une culture symbiotique de levures et de bactéries lactiques cultivées dans un milieu constitué de farine et d'eau. Quelques boissons au yaourt contiennent des lactobacilles comme complément alimentaire. Le kimchi coréen est également fabriqué en utilisant des techniques de fermentation par l'acide lactique. Beaucoup de lactobacilles ont ce caractère exceptionnel parmi les êtres vivants qu'ils n'exigent pas de fer pour leur croissance et qu'ils ont une tolérance très haute envers l'eau oxygénée.
Les fromages
Lors de la fabrication du fromage, les lactobacilles[9] interviennent au niveau de la coagulation du lait en produisant une acidification modérée du milieu puis à l'étape de l'affinage en contribuant aux qualités organoleptiques du produit. Ils sont particulièrement abondants dans les fromages à pâte pressée cuite où ils sont apportés par les levains (contenant Lactobacillus helveticus, Lactobacillus delbrueckii subsp. lactis, Lactobacillus delbrueckii subsp. bulgaricus). Les lactobacilles thermophiles libèrent des protéases qui se traduisent en fin d'affinage par une présence importante de petits peptides et d'acides aminés libres. Pour faire du yaourt, deux types de bactéries lactiques spécifiques sont requises dont un lactobacille (Lactobacillus delbrueckii subsp. bulgaricus et Streptococcus thermophilus).
Le pain
Les microbiotes de la farine de blé et des pâtes à pain sont riches en lactobacilles[10]. La farine contient des bactéries homofermentaires (Lb. coryniformis, Lb. curvatus, Lb. plantarum, Lb. salivarius) et hétérofermentaires (Lactobacillus brevis, Lb. fermentum). Le levain naturel servant à fabriquer le pain au levain est obtenu par fermentation spontanée de farine et d'eau. Son microbiote contient bien sûr un grand nombre de lactobacilles[11]. Les levain traditionnels sont en général dominés[12] par Lactobacillus sanfranciscensis, Lb. brevis et Lb. plantarum. Dans un levain naturel français, ce sont Lactobacillus acetotolerans et Lactobacillus panis qui dominent[13].
Le vin
Après la fermentation alcoolique du vin, il se produit une seconde fermentation dite malolactique qui contribue à désacidifier le vin et donc à lui donner souplesse et rondeur. Elle est assurée par une bactérie lactique Œnococcus œni qui transforme l'acide malique en acide lactique (moins fort) et en CO2. Pour les vins au pH supérieur 3,5, certains lactobacilles (comme Lactobacillus plantarum) sont aptes à produire cette fermentation malolactique[14]. Les principaux lactobacilles des moûts de raisin et des vins sont des hétérofermentaires comme Lactobacillus casei, Lb. plantarum, Lb. brevis, Lb. hilgardii.
Fermentation d'aliments courants en saumure
Dans la fermentation de produits en saumure de type choucroute, cornichons, olives, saucissons, on trouve une séquence de flores bactériennes souvent lorsque le milieu s'est acidifié par l'intervention des lactobacilles. La choucroute s'obtient par salage et fermentation du chou. La fermentation lactique est due au départ à Leuconostoc mesenteroides. Lorsque l'acidité atteint 1 %, la fermentation se poursuit grâce à Lactobacillus plantarum puis à partir de 2 %, par Lactobacillus brevis, Lb. curvatus, Lb. sakei etc.[2] La maturation des saucissons secs passe par une série d'étapes[2] : les bactéries gram positives disparaissent rapidement pour laisser la place aux microcoques, streptocoques et pédiocoques, puis viennent les lactobacilles (Lactobacillus Sakei (en)). Ces bactéries agissent à l'intérieur du saucisson en consommant les sucres, réduisant les nitrates en nitrites et en donnant des arômes. En surface, ce sont plutôt des levures et des moisissures qui contribuent à la protection du produit.
Altération des aliments
Dans les viandes, les lactobacilles provoquent un verdissement par action de H2O2 qui transforme l'hémoglobine en choléglobine[2]. L'altération de la bière peut être provoquée par Lactobacillus brevis, Lb. casei et plantarum. La même action défavorable se retrouve dans les produits issus de la fermentation de céréales (whisky) ou de fruits (cidre). De même, les jus sucrés sont altérés par Lb. casei et plantarum.
Intérêt probiotique
Un exemple type de probiotique est le kéfir. Beaucoup d'études sont menées sur les souches de lactobacilles susceptibles d'avoir un effet bénéfique sur la santé, en améliorant les propriétés du microbiote intestinal humain ou animal[15]. Les lactobacilles considérés comme des probiotiques sont d'après Hopzapfel et als (1998) : L. acidophilus, L. amylovorus, L. casei, L. crispatus, L. delbruckii subsp. bulgaricus, L. gallinarum, L. gasseri, L. johnsonii, L. paracasei, L. plantarum, L. reuteri, L. rhamnosus[16].
Des études récentes montrent, sur modèle animal, le microbiote des individus souffrant de troubles dépressifs majeurs produit peu de précurseurs de la sérotonine, compromettant ainsi l'efficacité d’antidépresseurs comme la fluoxetine[17],[18]. Selon une autre étude, le stress chronique, l'alimentation et le microbiote intestinal génèrent une « boucle d'anticipation pathologique » qui contribue au comportement dépressif via le système endocannabinoïde (en) (eCB) central. Les effets indésirables du microbiote ont pu être atténués en agissant sur l'eCB central ou par complémentation avec Lactobacillus plantarum[19].
Liste des espèces
Fin 2013, la liste selon LPS (EUZÉBY) comporte 192 espèces de lactobacilles et beaucoup d'entre elles sont de création récente, comme leur date de création l'indique.
- L. acetotolerans (1986)
- L. acidifarinæ (2005)
- L. acidipiscis (2000)
- L. acidophilus (1970, 1980)
- L. agilis (1982)
- L. algidus (2000)
- L. alimentarius (1983)
- L. amylolyticus (1999)
- L. amylophilus (1981)
- L. amylotrophicus (2006)
- L. amylovorus (1981)
- L. animalis (1983)
- L. antri (2005)
- L. apodemi (2006)
- L. aquaticus (2009)
- L. arizonensis (2000)
→ L. plantarum - L. aviarius (1985)
- L. bavaricus (1980)
→ L. sakei - L. bifermentans (1983)
- L. bobalius (2008)
→ L. paralimentarius - L. brantæ (2012)
- L. brevis (1934)
- L. buchneri (1923)
- L. cacaonum (2009)
- L. camelliæ (2007)
- L. capillatus (2008)
- L. casei (1971)
- L. catenaformis (1970)
→ Eggerthia catenaformis - L. ceti (2008)
- L. coleohominis (2001)
- L. collinoides (1972)
- L. composti (2007)
- L. concavus (2005)
- L. coryniformis (1965)
- L. crispatus (1970)
- L. crustorum (2007)
- L. curieæ (2013)
- L. curvatus (1965)
- L. delbrueckii subsp. delbrueckii
- L. delbrueckii subsp. indicus
- L. delbrueckii subsp. bulgaricus
- L. delbrueckii subsp. lactis
- L. delbrueckii subsp. sunkii (2012)
- L. dextrinicus (2009)
- L. diolivorans (2002)
- L. equi (2002)
- L. equicursoris (2010)
- L. equigenerosi (2008)
- L. fabifermentans (2009)
- L. farraginis (2007)
- L. farciminis (1983)
- L. fermentum (1901)
- L. floricola (2011)
- L. florum (2010)
- L. fornicalis (2000)
- L. fructivorans (1934)
- L. frumenti (2000)
- L. fuchuensis (2002)
- L. fusaii (2012)
- L. gallinarum (1992)
- L. gasseri (1980)
- L. gastricus (2005)
- L. ghanensis (2007)
- L. gigeriorum (2012)
- L. graminis (1989)
- L. hammesii (2005)
- L. hamsteri (1988)
- L. harbinensis (2006)
- L. hayakitensis (2007)
- L. helveticus (1925)
- L. hilgardii (1936)
- L. hokkaidonensis (2013)
- L. hominis (2013)
- L. homohiochii (1957)
- L. hordei (2008)
- L. iners (1999)
- L. ingluviei (2003)
- L. intestinalis (1990)
- L. jensenii (1970)
- L. johnsonii (1992)
- L. kalixensis (2005)
- L. kefiranofaciens (1988)
- L. kefirgranum (1994)
→ L. kefiranofaciens subsp. kefirgranum - L. kefiri (1983)
- L. kimchicus (2011)
- Lactobacillus kimchiensis (2013)
- L. kimchii (2000)
- L. kisonensis (2009)
- L. kitasatonis (2003)
- L. koreensis (2011)
- L. kunkeei (1998)
- L. leichmannii (1923)
- L. lindneri (1997)
- L. malefermentans (1989)
- L. mali (1970)
- L. manihotivorans (1998)
- L. mindensis (2003)
- L. mucosæ (2000)
- L. murinus (1982)
- L. nagelii (2000)
- L. namurensis (2007)
- L. nantensis (2006)
- L. nasuensis (2012)
- L. odoratitofui (2010)
- L. œni (2009)
- L. oligofermentans (2005)
- L. oris (1988)
- L. oryzæ (2013)
- L. otakiensis (2009)
- L. ozensis (2011)
- L. panis (1996)
- L. pantheris (2002)
- L. parabrevis (2006)
- L. parabuchneri (1989)
- L. paracasei (1989)
- L. paracollinoides (2004)
- L. parafarraginis (2007)
- L. parakefiri (1994)
- L. paralimentarius (1999)
- L. paraplantarum (1996)
- L. pasteurii (2013)
- L. paucivorans (2010)
- L. pentosus (1987)
- L. perolens (2000)
- L. plantarum (1923)
- L. pobuzihii (2010)
- L. pontis (1994)
- L. porcinæ (2013)
- L. psittaci (2001)
- L. rapi (2009)
- L. rennini (2006)
- L. reuteri (1982)
- L. rhamnosus (1989)
- L. rimæ (1991)
- L. rogosæ (1974)
- L. rossiæ (2005)
- L. ruminis (1973)
- L. særimneri (2004)
- L. sakei (1934)
- L. salivarius (1953)
- L. sanfranciscensis (1984)
- L. saniviri (2012)
- L. satsumensis (2005)
- L. secaliphilus (2007)
- L. selangorensis (2011)
- L. senioris (2012)
- L. sharpeæ (1982)
- L. siliginis (2006)
- L. similis (2010)
- L. sobrius (2006)
- L. spicheri (2004)
- L. sucicola (2009)
- L. suebicus (1989)
- L. sunkii (2009)
- L. suntoryeus (2005)
- L. taiwanensis (2009)
- L. thailandensis (2007)
- L. tucceti (2009)
- L. uli (1991)
- L. ultunensis (2005)
- L. uvarum (2009)
- L. vaccinostercus (1983)
- L. vaginalis (1989)
- L. versmoldensis (2003)
- L. vini (2006)
- L. vitulinus (1973)
- L. xiangfangensis (2012)
- L. yonginensis (2013)
- L. zeæ (1996)
- L. zymæ (2005)
- L. zymæ
Liens externes
- (en) Référence NCBI : Lactobacillus (taxons inclus).
- LPSN Euzéby.
- « La séquence du génome du lactobacille du yaourt est dévoilée », Proc. Natl. Acad. Sci., no 13, p. 9274-9279.
Notes
- Les bactéries du microbiote intestinal sont classées en autochtones, allochtones et xénochtones (Savage, 1977). Les bactéries autochtones sont attachées à la surface de l'épithélium intestinal ou adhèrent au mucus. Ce sont les premières colonisatrices du tractus digestif. Les bactéries allochtones sont localisées dans la lumière digestive, elles apparaissent plus tard et trouvant des biotopes déjà occupés, elles ne font que passer (Lamine, 2004).
Références
- W. H. Holzapfel et B. J. Wood, The Genera of Lactic Acid Bacteria, 2, Springer-Verlag, 1st ed. 1995 (2012), 411 p. (ISBN 978-2-89448-084-7 et 2-89448-084-9, lire en ligne), « The genus Lactobacillus par W. P. Hammes, R. F. Vogel ».
- J.-P. Guiraud, Microbiologie alimentaire, Dunod, .
- Antonia Suau, « Direct Analysis of Genes Encoding 16S rRNA from Complex Communities Reveals Many Novel Molecular Species within the Human Gut », Applied and Environmental Microbiology, vol. 65, no 11, , p. 4799-4807 (ISSN 0099-2240 et 1098-5336, lire en ligne).
- Florence Lamine, Lactobacillus farciminis une bactérie produisant du monoxyde d'azote dans le tube digestif : mise en évidence de potentialité thérapeutiques, thèse, Institut national polytechnique de Toulouse, .
- Ann M. O'Hara, « The gut flora as a forgotten organ », EMBO Reports, vol. 7, no 7, , p. 688-693 (ISSN 1469-221X, PMCID PMC1500832, DOI 10.1038/sj.embor.7400731, lire en ligne).
- Jens Walter, « Ecological Role of Lactobacilli in the Gastrointestinal Tract: Implications for Fundamental and Biomedical Research », Applied and Environmental Microbiology, vol. 74, no 16, , p. 4985-4996 (ISSN 0099-2240 et 1098-5336, DOI 10.1128/AEM.00753-08, lire en ligne)0
- Jens Walter, « Detection of Lactobacillus, Pediococcus, Leuconostoc, and Weissella Species in Human Feces by Using Group-Specific PCR Primers and Denaturing Gradient Gel Electrophoresis », Applied and Environmental Microbiology, vol. 67, no 6, , p. 2578-2585 (ISSN 0099-2240 et 1098-5336, DOI 10.1128/AEM.67.6.2578-2585.2001, lire en ligne).
- C. Badet, « Ecology of Lactobacilli in the Oral Cavity: A Review of Literature », The Open Microbiology Journal, vol. 2, , p. 38-48 (ISSN 1874-2858, DOI 10.2174/1874285800802010038, lire en ligne).
- André Eck, Jean-Claude Gillis (coord.), Le fromage, De la science à l'assurance-qualité, TEC&DOC, lavoisier, , 891 p. (ISBN 978-2-7430-0891-8), chap. 10 (« Les phénomènes microbiens par C. Choisy, Desmazeaud, Gueguen, Lenoir, Schmidt, Tourneur »).
- Luc De Vuyst, « The sourdough microflora: biodiversity and metabolic interactions », Trends in Food Science & Technology, vol. 16, nos 1-3, , p. 43-56 (ISSN 0924-2244, DOI 10.1016/j.tifs.2004.02.012, lire en ligne, consulté le ).
- (en) Karel Kulp, Handbook of Dough Fermentations, New York/Basel, Marcel Dekker Inc., , 304 p. (ISBN 0-8247-4264-8).
- (de) M. J. Brandt et M. G. Gänzle, Handbuch Sauerteig., Behrs Verlag, , 92-95 p..
- Annabelle Vera, « Description of a French natural wheat sourdough over 10 consecutive days focussing on the lactobacilli present in the microbiota », Antonie van Leeuwenhoek, vol. 101, no 2, , p. 369-377 (ISSN 1572-9699, DOI 10.1007/s10482-011-9642-6).
- Pascal Ribéreau-Gayon, D. Dubourdieu, B. Donèche, A. Lonvaud, Traité d'œnologie, tome 1, Dunod, 2012 (6e éd.), 676 p..
- W. H. Holzapfel, « Taxonomy and important features of probiotic microorganisms in food and nutrition », The American journal of clinical nutrition, vol. 73, no 2 Suppl, , p. 365S-373S (ISSN 0002-9165).
- W. H. Holzapfel, « Overview of gut flora and probiotics », International journal of food microbiology, vol. 41, no 2, , p. 85-101 (ISSN 0168-1605).
- Pierre-Marie Lledo et Gérard Eberl, « Dépression et microbiote : « Une grande révolution médicale arrive » », sur Polytechnique insights, (consulté le ).
- (en) Eleni Siopi et al., « Changes in Gut Microbiota by Chronic Stress Impair the Efficacy of Fluoxetine », Cell Reports, vol. 30, no 11, , p. 3682-3690 (DOI 10.1016/j.celrep.2020.02.099, lire en ligne, consulté le ).
- (en) Grégoire Chevalier et al., « Effect of gut microbiota on depressive-like behaviors in mice is mediated by the endocannabinoid system », Cell Reports, (DOI 10.1038/s41467-020-19931-2, lire en ligne, consulté le ).
- Portail de la microbiologie