Désamination
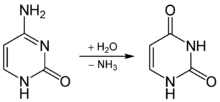
La désamination est une réaction chimique au cours de laquelle une molécule perd un groupement amine. Les enzymes catalysant cette réaction sont appelées désaminases.
Dans le corps humain, la désamination a lieu principalement dans le foie, néanmoins on trouve également la désamination du glutamate dans les reins. En situation d'apport excessif de protéines, la désamination est utilisée pour cliver les acides aminés afin d'apporter de l'énergie. Le groupe amine est retiré de l'acide aminé et converti en ammoniac. La partie restante de l'acide aminé est composée principalement de carbone et d'hydrogène, et est recyclée ou oxydée pour donner de l'énergie. L'ammoniac étant toxique pour l'organisme humain, des enzymes le convertissent en urée ou acide urique en ajoutant des molécules de dioxyde de carbone (ce qui n'est pas considéré comme un processus de désamination) dans le cycle de l'urée, qui a également lieu dans le foie. L'urée et l'acide urique peuvent diffuser sans encombre dans le sang pour finir par être excrétés dans l'urine.
Réactions de désamination dans l'ADN
Cytosine et 5-méthylcytosine
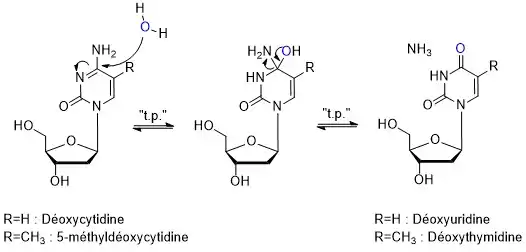
La désamination spontanée correspond à l'hydrolyse de la cytosine en uracile ou de la 5-méthylcytosine en thymine, processus au cours duquel de l'ammoniac est libérée. Cela peut se produire in vitro par l'usage de bisulfite, qui désamine la cytosine mais pas la 5-méthylcytosine. Cette propriété a permis aux chercheurs de séquencer de l'ADN méthylé afin de distinguer la cytosine non méthylée (correspondant à l'uracile) de la cytosine méthylée (non modifiée).
Dans l'ADN, cette désamination spontanée est corrigée par le retrait d'uracile (produit de la désamination de la cytosine et ne faisant pas partie de l'ADN) par l'uracile-ADN glycosylase, générant un site abasique (AP). Le site abasique résultant est ensuite reconnu par des enzymes (AP endonucléases) qui rompent une liaison phosphodiester dans l'ADN, permettant la réparation de la lésion résultante via son remplacement par une autre cytosine. Une ADN polymérase peut effectuer ce remplacement via une traduction de coupure ("nick translation" en anglais), une réaction d'excision terminale de par son activité 5' → 3' exonucléase, suivie d'une réaction de "remplissage" de par son activité polymérase. Ensuite, une ligase forme une liaison phosphodiester afin de sceller le produit résultant, correspondant à une nouvelle cytosine bien appariée. [Voir : Réparation par excision de base.]
La désamination spontanée de la 5-méthylcytosine a pour produits de la thymine et de l'ammoniac. Il s'agit de la mutation d'un nucléotide simple la plus commune. Dans l'ADN, si cette réaction est détectée avant le passage de la fourche de réplication, elle peut être corrigée par la thymine-ADN glycosylase, qui retire la base thymine dans une association G/T mal assortie. Cela laisse un site abasique qui est réparé par les AP endonucléases et la polymérase, de la même façon qu'avec l'uracile-ADN glycosylase[1].
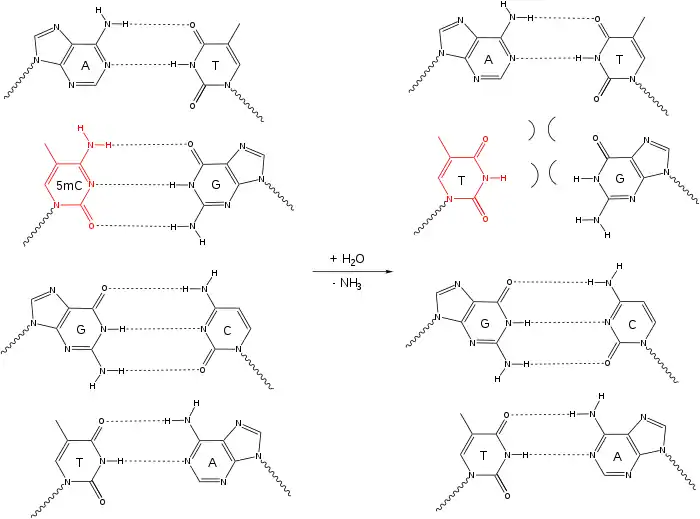
Les dinucléotides CpG, sites où sont retrouvées la majorité des cytosines méthylées, sont des régions qui manifestent une propension élevée de mutations chez la souris, le rat et l'humain[2]. Ainsi, une grande proportion des transitions de l'appariement GC vers l'appariement AT sont retrouvées aux sites CpG[3]. Il est présumé que la désamination de la 5-méthylcytosine pourrait être responsable de ce phénomène[4]. En effet, si cette dernière désamine lors de la division cellulaire sans être réparée, le brin d'ADN mère contiendra une thymine au lieu de la 5-méthylcytosine, ce qui donnera un appariement avec une adénine, générant par conséquent la mutation ponctuelle sur le brin d'ADN fille correspondant. Donc, une transition GC vers AT.
Dans l'exemple ci-dessous, la séquence 5'-ACGT-3' illustre le phénomène de désamination au sein d'une molécule d'ADN double-brin. Si la 5-méthylcytosine (5mC) du dinucléotide CpG subit une désamination, la thymine ainsi générée n'est plus complémentaire à la guanine attenante.
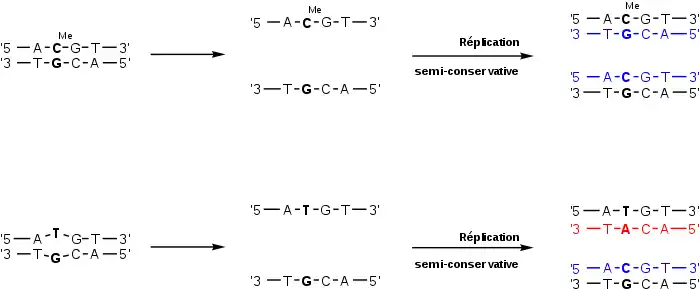
Si une telle transition apparaît dans une cellule sans que cela soit corrigé et que la division cellulaire a lieu, une mutation GC → AT apparaît dans la cellule fille possédant le brin d'ADN mère sur lequel s'est produit la transition. En effet, lors de la division cellulaire, les deux brins d'ADN sont séparés puis répliqués, ce qui double le matériel génétique afin d'obtenir deux cellules filles. De ce fait, chacune des deux cellules filles détient un brin d'ADN provenant de la cellule mère (en noir ci-dessous) et un brin nouvellement synthétisé (en couleur), un processus dit « semi-conservatif ». Le premier exemple montre que les deux cellules filles détiennent la même séquence génétique, tandis que le deuxième exemple montre une situation dans laquelle une mutation est apparue à la suite de la transition 5mC → T sur le brin du haut.
Guanine
La désamination de la guanine résulte en la formation de xanthine. La xanthine, de façon analogue à la tautomère énol de la guanine, s'apparie de façon sélective avec la thymine au lieu de la cytosine. Cela mène à une mutation transitoire post-réplication, où la paire de bases originelle G-C se transforme en paire de bases A-T. La correction de cette mutation implique l'utilisation d'une alkyladenine glycosylase (Aag) lors de la réparation par excision de base.
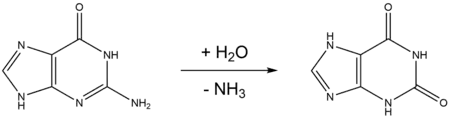
Adénine
La désamination de l'adénine résulte en la formation d'hypoxanthine, entrainant ainsi la modification d'une adénosine en une inosine [5]. L'hypoxanthine, de façon analogue à la tautomère imine de l'adénine, s'apparie de façon sélective avec la cytosine au lieu de la thymine. Cela mène à une mutation transitoire post-réplication, où la paire de bases originelle A-T se transforme en paire de bases G-C.

Informations complémentaires
Il est à noter que la présence des sites AP dans la séquence génétique sont dus, entre autres, au fait que l'efficacité du système REB (réparation par excision de base) tend à baisser avec l'âge[6].
Par ailleurs, la désamination fait partie des causes endogènes de production de défauts dans la chaîne d'ADN : chez les mammifères, 100 à 500 défauts de désamination sont produits par jour et par cellule[7].
Protéines supplémentaires accomplissant cette fonction
- APOBEC1
- APOBEC3A-H, APOBEC3G - affecte le VIH
- Cytidine désaminase d'activation induite (AICDA)
- Cytodine désaminase de mononucléotides (CDA)
- Adénosine désaminase agissant sur l'ARNt (ADAT)
- Adénosine désaminase agissant sur l'ARNdb (ADAR)
- Adénosine désaminase agissant sur des mononucléotides (ADA)
- Guanine désaminase (GDA)
Notes et références
- (en) Petra Neddermann, Paola Gallinari, Teresa Lettieri et Daniel Schmid, « Cloning and Expression of Human G/T Mismatch-specific Thymine-DNA Glycosylase », Journal of Biological Chemistry, vol. 271, no 22, , p. 12767–12774 (ISSN 0021-9258 et 1083-351X, PMID 8662714, DOI 10.1074/jbc.271.22.12767, lire en ligne, consulté le )
- (en) Shulin Zhang, Barry W. Glickman et Johan G. de Boer, « Spontaneous mutation of the lacI transgene in rodents: Absence of species, strain, and insertion-site influence », Environmental and Molecular Mutagenesis, vol. 37, no 2, , p. 141–146 (ISSN 1098-2280, DOI 10.1002/em.1021, lire en ligne, consulté le )
- Guru S. Madugundu, Jean Cadet et J. Richard Wagner, « Hydroxyl-radical-induced oxidation of 5-methylcytosine in isolated and cellular DNA », Nucleic Acids Research, vol. 42, no 11, , p. 7450–7460 (ISSN 0305-1048, PMID 24852253, PMCID PMC4066766, DOI 10.1093/nar/gku334, lire en ligne, consulté le )
- (en) C. P. Walsh et G. L. Xu, DNA Methylation: Basic Mechanisms, Springer, Berlin, Heidelberg, coll. « Current Topics in Microbiology and Immunology », (ISBN 3540313907, DOI 10.1007/3-540-31390-7_11, lire en ligne), p. 283–315
- Cyril X. George, Lijo John et Charles E. Samuel, « An RNA Editor, Adenosine Deaminase Acting on Double-Stranded RNA (ADAR1) », Journal of Interferon & Cytokine Research, vol. 34, no 6, , p. 437–446 (ISSN 1079-9907, PMID 24905200, PMCID PMC4046350, DOI 10.1089/jir.2014.0001, lire en ligne, consulté le )
- Benjamin P. Best, « Nuclear DNA damage as a direct cause of aging », Rejuvenation Research, vol. 12, no 3, , p. 199–208 (ISSN 1557-8577, PMID 19594328, DOI 10.1089/rej.2009.0847, lire en ligne, consulté le )
- (en) Front Matter | Health Risks from Exposure to Low Levels of Ionizing Radiation: BEIR VII Phase 2 | The National Academies Press (DOI 10.17226/11340, lire en ligne)
- Portail de la chimie
- Portail de la biochimie
- Portail de la microbiologie