Code génétique
Le code génétique est l'ensemble des règles permettant de traduire les informations contenues dans le génome des cellules vivantes afin de synthétiser les protéines. Au sens large, il établit la correspondance entre le génotype et le phénotype d'un organisme. Ce code repose notamment sur la correspondance entre, d'une part, des triplets de nucléotides, appelés codons, sur l'ARN messager et, d'autre part, les acides aminés protéinogènes incorporés dans les protéines synthétisées lors de la phase de traduction de l'ARN messager par les ribosomes.
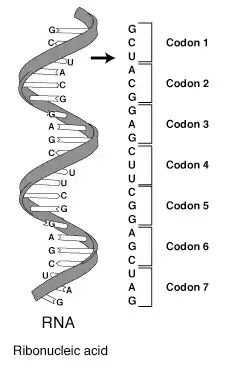
À quelques exceptions près[1], chaque codon correspond à un seul et unique acide aminé protéinogène. Dans la mesure où l'information génétique est codée exactement de la même façon dans les gènes de la très grande majorité des différentes espèces vivantes, ce code génétique spécifique est généralement désigné comme code génétique standard, ou canonique, voire tout simplement comme « Le » code génétique ; il existe cependant un certain nombre de variantes à ce code génétique, mais qui restent limitées en général à quelques codons. De telles variantes existent par exemple au sein même des cellules humaines entre leur cytosol et leurs mitochondries.
La correspondance entre codons d'ARN messager et acides aminés protéinogènes est généralement présentée sous forme de tableaux associant chacun des 64 codons, ou triplets de quatre bases nucléiques possibles (43 = 64), avec l'un des 22 acides aminés protéinogènes.
Par extension, et de façon impropre, le grand public appelle parfois « code génétique » ce qui est en fait le génotype d'une cellule, c'est-à-dire l'ensemble de ses gènes.
Caractéristiques principales
Lors de l'expression des protéines à partir du génome, des segments de l'ADN génomique sont transcrits en ARN messager. Cet ARN messager (ou ARNm) contient des régions non codantes, qui ne sont pas traduites en protéines, et une ou plusieurs régions codantes, qui sont traduites par les ribosome pour produire une ou plusieurs protéines. L'ARNm est composé de l'enchaînement de quatre types de bases nucléiques, A, C, G et U, qui constituent les « lettres » avec lesquelles est écrit le code génétique. Ce dernier est constitué de « mots » de 3 lettres (nucléotides) appelés codons. Dans les régions codantes de l'ARN messager, chaque codon est traduit en l'un des 22 acides aminés protéinogènes dans la protéine à synthétiser.
Le nombre de mots de trois lettres pris dans un alphabet de quatre lettres étant de 43, le code génétique comporte 64 codons différents, codant directement 20 acides aminés dits "standards", ainsi que le signal de fin de la traduction, ce dernier étant codé par l'un des 3 codons-stop ou codons de terminaison. Deux acides aminés rares, la sélénocystéine et la pyrrolysine, sont insérés au niveau de certains codons-stop, dont le recodage en codons d'acides aminés intervient en présence de structures particulières de type tige-boucle, ou épingle à cheveux, induites par des séquences d'insertion spécifiques sur l'ARN messager.
Cadre de lecture
Un codon est défini par le premier nucléotide à partir duquel la traduction commence. Ainsi la chaîne GGGAAACCC peut être lue selon les codons GGG·AAA·CCC, GGA·AAC et GAA·ACC selon que l'on commence la lecture des codons à partir du premier, du deuxième ou du troisième nucléotide, respectivement. Toute séquence nucléotidique peut ainsi être lue selon trois cadres de lecture distincts, qui résultent en des traductions en acides aminés totalement différentes : dans notre exemple, on aurait respectivement les acides aminés Gly–Lys–Pro, Gly–Asn et Glu–Thr.
Dans les gènes, le cadre de lecture commence généralement avec un codon AUG codant la méthionine, ou la N-formylméthionine chez les bactéries et dans les mitochondries et les chloroplastes des eucaryotes.
Codons d'initiation, de terminaison
La traduction génétique par les ribosomes commence avec un codon d'initiation, parfois appelé codon de démarrage. Contrairement aux codons-stop, le codon d'initiation seul ne suffit pas à commencer la traduction. Le site de fixation du ribosome (RBS) chez les procaryotes et les facteurs d'initiation chez les procaryotes et les eucaryotes sont indispensables à l'amorçage de la traduction. Le codon d'initiation le plus courant est AUG, correspondant à la méthionine ou, chez les bactéries, à la N-formylméthionine. GUG et UUG, qui correspondent respectivement à la valine et à la leucine dans le code génétique standard, peuvent également être des codons d'initiation chez certains organismes, étant dans ce cas interprétés comme des codons de méthionine ou de N-formylméthionine[2].
Les trois codons-stop UAG, UGA et UAA, se sont vus attribuer des noms lors de leur découverte, respectivement ambre, opale et ocre[3]. Ils sont également appelés codons non-sens ou codons de terminaison. Ils provoquent l'arrêt du ribosome et la libération de la chaîne polypeptidique néoformée par absence d'ARN de transfert ayant des anticodons adaptés (il n'y a pas d'acide aminé correspondant aux triplets UAG, UGA et UAA), ce qui provoque la liaison d'un facteur de terminaison[4].
Effet des mutations
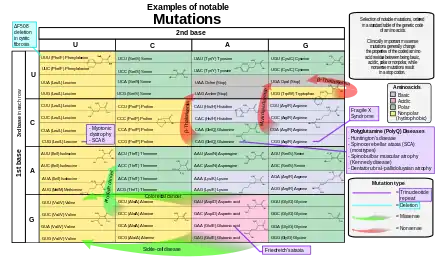
Lors de la réplication de l'ADN, des erreurs de transcription sont susceptibles de se produire lors de la polymérisation du second brin d'ADN par l'ADN polymérase. Ces erreurs, appelées mutations, peuvent avoir des conséquences sur le phénotype d'un être vivant, notamment si elles surviennent dans les régions codantes d'un gène. Le taux d'erreur est généralement très faible, de l'ordre d'une erreur de réplication sur dix à cent millions de bases répliquées, grâce à la fonction de relecture et correction d'épreuve (proofreading) des ADN polymérases[5].
Les mutations faux-sens et les mutations non-sens sont des exemples de mutations ponctuelles, susceptibles de provoquer des maladies génétiques telles que, respectivement, la drépanocytose et la thalassémie[6],[7]. Les mutations faux-sens qui ont un impact physiologique important sont celles qui conduisent à changer la nature physicochimique — par exemple l'encombrement stérique, la nature hydrophile ou hydrophobe, la charge électrique, la nature acide ou basique — d'un résidu d'acide aminé important pour la fonction de la protéine modifiée. Les mutations non-sens conduisent à introduire prématurément un codon-stop dans la séquence de la protéine à transcrire, qui s'en trouve tronquée et donc la fonction physiologique dans les tissus se trouve généralement altérée.
Les mutations qui affectent la transcription par indels — insertions et délétions — d'un nombre de nucléotides qui n'est pas multiple de 3 correspondent à un décalage du cadre de lecture. De telles mutations conduisent généralement à un polypeptide totalement différent de l'original, aussi bien dans la séquence des résidus d'acides aminés traduits que dans la longueur de la chaîne polypeptidique produite, puisque la position des codons-stop est généralement modifiée lors d'une telle mutation[8]. Ces mutations sont susceptibles de rendre les protéines résultantes inopérantes, ce qui les rend très rares dans les séquences encodant des protéines car elles sont souvent incompatibles avec la survie de l'organisme affecté[9]. Lorsqu'elles surviennent, elles peuvent provoquer des maladies génétiques graves telles que la maladie de Tay-Sachs.
Si la grande majorité des mutations qui ont un impact sur la séquence des protéines sont délétères ou sans conséquences, certaines peuvent avoir des effets bénéfiques[10]. Certaines de ces mutations peuvent par exemple permettre aux organismes chez lesquels elles surviennent de supporter des conditions de stress environnemental mieux que la forme sauvage, ou de se multiplier plus rapidement. Ces mutations se trouvent alors favorisées par sélection naturelle. Les virus à ARN présentent un taux de mutation élevé[11], ce qui constitue pour eux un avantage leur permettant d'évoluer continuellement et de se soustraire au système immunitaire de leur hôte[12]. Au sein de grandes populations d'organismes qui se reproduisent de façon asexuée, par exemple chez E. coli, plusieurs mutations bénéfiques peuvent survenir en même temps ; ce phénomène est appelé interférence clonale et se manifeste par la compétition entre ces différentes mutations[13], conduisant souvent à la généralisation de l'une d'elles au détriment des autres.
Dégénérescence du code génétique
Le fait que les 64 codons codent seulement 22 acides aminés protéinogènes, plus les codons de terminaison, conduit à de très nombreuses redondances. Ceci fait qu'un acide aminé standard est codé en moyenne par trois codons distincts — jusqu'à six codons différents. On parle de codons synonymes. Parmi les 20 acides aminés standards, seuls la méthionine et le tryptophane ne sont codés que par un codon, tandis que l'asparagine, l'aspartate, la cystéine, le glutamate, la glutamine, l'histidine, la lysine, la phénylalanine et la tyrosine sont codés par deux codons distincts, l'isoleucine et la terminaison de traduction sont codées par trois codons distincts, la thréonine, la proline, l'alanine, la glycine et la valine sont codées par quatre codons distincts, et l'arginine, la leucine et la sérine sont codées par six codons. Il existe donc souvent plusieurs ARN de transfert associés à un même acide aminé, capables de se lier aux différents triplets dégénérés de nucléotides sur l'ARN. On parle alors d'ARNt isoaccepteurs, car ils acceptent le même acide aminé.
L'utilisation par un organisme donné des différents codons synonymes pour un acide aminé n'est pas aléatoire. On observe en général ce qu'on appelle un biais d'usage du code. La cellule exprime en général des préférences assez marquées dans le choix des codons synonymes, ainsi par exemple, le codon AUA qui code l'isoleucine est largement évité chez l'homme comme chez Escherichia coli, par rapport aux deux autres codons synonymes AUU et AUC. Cette préférence d'usage des codons est très variable selon l'organisme, et dépend, au sein d'un même génome, de la fraction considérée (nucléaire, mitochondriale, chloroplastique). Elle est en revanche assez générale pour l'ensemble des gènes portés par la même fraction du génome.
Si le code génétique est dégénéré, il n'est en revanche pas ambigu : chaque codon ne spécifie normalement qu'un acide aminé et un seul. Chaque acide aminé standard est encodé en moyenne par trois codons différents, de sorte que, statistiquement, une mutation sur trois n'entraîne aucune modification de la protéine traduite : on dit alors qu'une telle mutation est silencieuse. Une conséquence pratique de cette dégénérescence est qu'une mutation sur le troisième nucléotide d'un codon n'engendre généralement qu'une mutation silencieuse ou bien la substitution d'un résidu par un autre présentant les mêmes propriétés hydrophiles ou hydrophobes, acides ou basiques, et de même encombrement stérique.
Prévalence des codons
On pourrait s'attendre à ce que les fréquences des codons synonymes pour un acide aminé donné soient équivalentes, mais au contraire les études constatent une prévalence de codons (en anglais : codon bias) qui tend à affecter la structure finale des protéines[14]. Cette prévalence connaîtrait par ailleurs une certaine variabilité entre lignées[15].
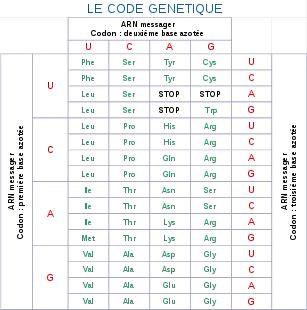
Table des codons d'ARN messager
C'est sur le code génétique que repose la biosynthèse des protéines. L'ADN est transcrit en ARN-messager (ARNm). Celui-ci est traduit par les ribosomes qui assemblent les acides aminés présents sur des ARN de transfert (ARNt). L'ARNt contient un « anti-codon », complémentaire d'un codon, et porte l'acide aminé correspondant au codon. L'estérification spécifique de l'acide aminé correspondant à un ARNt donné est réalisé par les aminoacyl-ARNt synthétases, une famille d'enzymes spécifiques chacune d'un acide aminé donné. Pendant la traduction, le ribosome lit l'ARNm codon par codon, met en relation un codon de l'ARNm avec l'anti-codon d'un ARNt et ajoute l'acide aminé porté par celui-ci à la protéine en cours de synthèse.
Le tableau suivant donne la signification standard de chaque codon de trois bases nucléiques d'ARN messager. Les principaux codages alternatifs sont indiqués après une barre oblique :
| 1re base |
2e base | 3e base | |||||||
|---|---|---|---|---|---|---|---|---|---|
| U | C | A | G | ||||||
| U | UUU | F Phe |
UCU | S Ser |
UAU | Y Tyr |
UGU | C Cys |
U |
| UUC | F Phe |
UCC | S Ser |
UAC | Y Tyr |
UGC | C Cys |
C | |
| UUA | L Leu |
UCA | S Ser |
UAA | Stop ocre | UGA | Stop opale / U Sec / W Trp |
A | |
| UUG | L Leu / initiation |
UCG | S Ser |
UAG | Stop ambre / O Pyl |
UGG | W Trp |
G | |
| C | CUU | L Leu |
CCU | P Pro |
CAU | H His |
CGU | R Arg |
U |
| CUC | L Leu |
CCC | P Pro |
CAC | H His |
CGC | R Arg |
C | |
| CUA | L Leu |
CCA | P Pro |
CAA | Q Gln |
CGA | R Arg |
A | |
| CUG | L Leu / initiation |
CCG | P Pro |
CAG | Q Gln |
CGG | R Arg |
G | |
| A | AUU | I Ile |
ACU | T Thr |
AAU | N Asn |
AGU | S Ser |
U |
| AUC | I Ile |
ACC | T Thr |
AAC | N Asn |
AGC | S Ser |
C | |
| AUA | I Ile |
ACA | T Thr |
AAA | K Lys |
AGA | R Arg |
A | |
| AUG | M Met & initiation |
ACG | T Thr |
AAG | K Lys |
AGG | R Arg |
G | |
| G | GUU | V Val |
GCU | A Ala |
GAU | D Asp |
GGU | G Gly |
U |
| GUC | V Val |
GCC | A Ala |
GAC | D Asp |
GGC | G Gly |
C | |
| GUA | V Val |
GCA | A Ala |
GAA | E Glu |
GGA | G Gly |
A | |
| GUG | V Val |
GCG | A Ala |
GAG | E Glu |
GGG | G Gly |
G | |
Une façon compacte de représenter la même information fait appel aux symboles à une lettre des acides aminés[16] :
Acide aminé : FFLLSSSSYY**CC*WLLLLPPPPHHQQRRRRIIIMTTTTNNKKSSRRVVVVAAAADDEEGGGG Initiation : ···M···············M···············M···························· 1re base : UUUUUUUUUUUUUUUUCCCCCCCCCCCCCCCCAAAAAAAAAAAAAAAAGGGGGGGGGGGGGGGG 2e base : UUUUCCCCAAAAGGGGUUUUCCCCAAAAGGGGUUUUCCCCAAAAGGGGUUUUCCCCAAAAGGGG 3e base : UCAGUCAGUCAGUCAGUCAGUCAGUCAGUCAGUCAGUCAGUCAGUCAGUCAGUCAGUCAGUCAG
- Tableau inverse
Comme chaque acide aminé d'une protéine est codé par un ou plusieurs codons, il est parfois utile de se référer au tableau suivant ; les principaux codages alternatifs sont indiqués en petits caractères entre parenthèses.
| Acide aminé | Codons | Compacté[17] | |||
|---|---|---|---|---|---|
| Alanine | A | Ala | GCU, GCC, GCA, GCG. | GCN | |
| Arginine | R | Arg | CGU, CGC, CGA, CGG ; AGA, AGG. | CGN, MGR | |
| Asparagine | N | Asn | AAU, AAC. | AAY | |
| Acide aspartique | D | Asp | GAU, GAC. | GAY | |
| Cystéine | C | Cys | UGU, UGC. | UGY | |
| Glutamine | Q | Gln | CAA, CAG. | CAR | |
| Acide glutamique | E | Glu | GAA, GAG. | GAR | |
| Glycine | G | Gly | GGU, GGC, GGA, GGG. | GGN | |
| Histidine | H | His | CAU, CAC. | CAY | |
| Isoleucine | I | Ile | AUU, AUC, AUA. | AUH | |
| Leucine | L | Leu | UUA, UUG ; CUU, CUC, CUA, CUG. | YUR, CUN | |
| Lysine | K | Lys | AAA, AAG. | AAR | |
| Méthionine | M | Met | AUG. | ||
| Phénylalanine | F | Phe | UUU, UUC. | UUY | |
| Proline | P | Pro | CCU, CCC, CCA, CCG. | CCN | |
| Pyrrolysine | O | Pyl | UAG, avant élément PYLIS. | ||
| Sélénocystéine | U | Sec | UGA, avec séquence SECIS. | ||
| Sérine | S | Ser | UCU, UCC, UCA, UCG ; AGU, AGC. | UCN, AGY | |
| Thréonine | T | Thr | ACU, ACC, ACA, ACG. | ACN | |
| Tryptophane | W | Trp | UGG. (UGA) | ||
| Tyrosine | Y | Tyr | UAU, UAC. | UAY | |
| Valine | V | Val | GUU, GUC, GUA, GUG. | GUN | |
| Initiation | AUG. (UUG, CUG) | ||||
| Terminaison | * | UAG, UAA ; UGA. | UAR, URA | ||
La région codante d'un ARNm se termine par un codon-stop. Il existe trois codons-stop (UAG, UAA et UGA) qui déclenchent l'arrêt de la traduction par le ribosome et la libération de la protéine terminée.
Variantes du code génétique
L'existence de variantes au code génétique a été mise en évidence en 1979 avec le code génétique des mitochondries humaines, et plus généralement celui des mitochondries de vertébrés :
Acide aminé : FFLLSSSSYY**CCWWLLLLPPPPHHQQRRRRIIMMTTTTNNKKSS**VVVVAAAADDEEGGGG Initiation : ································MMMM···············M············ 1re base : UUUUUUUUUUUUUUUUCCCCCCCCCCCCCCCCAAAAAAAAAAAAAAAAGGGGGGGGGGGGGGGG 2e base : UUUUCCCCAAAAGGGGUUUUCCCCAAAAGGGGUUUUCCCCAAAAGGGGUUUUCCCCAAAAGGGG 3e base : UCAGUCAGUCAGUCAGUCAGUCAGUCAGUCAGUCAGUCAGUCAGUCAGUCAGUCAGUCAGUCAG
De nombreuses autres variantes au code génétique ont été observées depuis lors[16], dont plusieurs variantes mitochondriales[18] et de légères variantes telles que la traduction du codon UGA par le tryptophane plutôt qu'un codon-stop chez Mycoplasma et la traduction du codon CUG par la sérine plutôt que la leucine chez certaines levures telles que Candida albicans[19],[20],[21]. Le tableau ci-dessous résume quelques variantes importantes du code génétique[16] :
| Codons d'ARN messager | UGA | CUU | CUC | CUA | CUG | GUG | CGA | CGC | AUU | AUC | AUA | AGA | AGG |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Code génétique standard | Term | Leu | Leu | Leu | Leu | Val | Arg | Arg | Ile | Ile | Ile | Arg | Arg |
| Mitochondries de vertébrés | Trp | Init | Init | Init | Term | Term | |||||||
| Mitochondries des ascidies | Trp | Init | Init | Init | Gly | Gly | |||||||
| Mitochondries de levures | Trp | Thr | Thr | Thr | Thr | Abs | Abs | Init | |||||
| Mitochondries d'invertébrés | Trp | Init | Init | Init | Ser | Ser | |||||||
| Bactéries, archées et plastes de plantes | Init | Init | Init | Init |
Dans la mesure où les virus se reproduisent en utilisant les ressources métaboliques — et donc le code génétique — de leurs hôtes, une variation du code génétique est susceptible d'avoir une incidence sur les protéines synthétisées et donc leur capacité à se reproduire ; certains virus, comme ceux du genre Totivirus (en), se sont ainsi adaptés aux variations du code génétique de leur hôte[22]. Chez les bactéries et les archées, GUG et UUG sont des codons d'initiation courants mais, dans certains cas rares, certaines protéines utilisent des codons d'initiations qui ne sont normalement pas ceux de ces espèces[16].
Certaines protéines utilisent des acides aminés non standards codés par des codons-stop en présence de séquences particulières sur l'ARN messager. Ainsi, le codon-stop UGA peut être recodé en sélénocystéine en présence d'un élément SECIS tandis que le codon-stop UAG peut être recodé en pyrrolysine en présence d'un élément PYLIS. Contrairement à la sélénocystéine, la pyrrolysine est liée à son ARN de transfert par une aminoacyl-ARNt synthétase dédiée[23]. Ces deux acides aminés non standards peuvent être présents chez le même organisme mais utilisent des modes d'expression différents[24]. Une archée telle que Acetohalobium arabaticum est capable, selon les conditions de son environnement, d'étendre son code génétique de 20 à 21 acides aminés en y incluant la pyrrolysine[25].
Toutes ces différences demeurent malgré tout marginales, et les codes génétiques des tous les organismes restent essentiellement très semblables : ils reposent sur des codons adjacents de trois nucléotides d'ARN messager lus toujours dans le même sens par des ribosomes qui assemblent des protéines à partir d'acides aminés protéinogènes selon une séquence déterminée par l'appariement des anticodons des ARN de transfert sur les codons d'ARN messager.
Théories relatives à l'origine et à l'évolution du code génétique
Il existe pas moins de 1,5×1084 possibilités de coder 21 éléments d'information (les 20 acides aminés standards + la fin de la traduction) par 64 codons[26], chiffre qui correspond au nombre de combinaisons possibles permettant d'associer 64 codons à 21 éléments d'information de telle sorte que tout codon et tout élément d'information soit associé à au moins une combinaison. Malgré ce nombre astronomiquement grand, tous les codes génétiques de toutes les formes de vies connues sont quasiment identiques, se limitant à un petit nombre de variations mineures. Les raisons d'une telle homogénéité universellement observée demeurent fondamentalement inconnues, bien que diverses hypothèses aient été formulées pour expliquer cette situation. Elles sont essentiellement de quatre types[27] :
- Les acides aminés et l'ARN présenteraient des affinités chimiques réciproques à l'origine de certaines propriétés du code génétique. En particulier, des expériences avec des aptamères ont mis en évidence que certains acides aminés présentent une affinité chimique spécifique pour les triplets de bases nucléiques qui les encodent[28]. D'autres expériences ont mis en évidence le fait que, sur huit acides aminés étudiés, six présentent des associations ARN-acide aminés[29],[30]. Enfin, les ARN de transfert semblent être apparus avant leurs aminoacyl-ARNt synthétases associées[31].
- Le code génétique actuel aurait été plus simple par le passé, avec incorporation progressive de nouveaux acides aminés, présents par exemple comme métabolites dans les cellules. Certaines études ont ainsi tenté de déterminer, par une extrapolation statistique, les acides aminés présents chez le dernier ancêtre commun universel[32], tandis que d'autres études, bien plus controversées, ont proposé des mécanismes expliquant comment le code génétique aurait pu incorporer progressivement un nombre croissant d'acides aminés différents[33],[34].
- La sélection naturelle aurait conduit à retenir un code génétique qui minimise les effets des mutations génétiques[35]. Il est également possible que des codons plus longs (par exemple, des quadruplets de nucléotides et non des triplets comme aujourd'hui) aient existé par le passé[36], ce qui aurait présenté un plus haut niveau de redondances et aurait donc été moins sujet aux erreurs que les triplets. Cette propriété aurait permis de préserver un décodage fidèle avant que les cellules disposent de la machinerie de traduction génétique complexe que sont les ribosomes.
- La représentation du processus de décodage de l'information génétique en acides aminés à travers la théorie de l'information conduit à des modèles de flux d'information sujets aux erreurs[37]. Le bruit propre à ce flux de données pose un problème fondamental aux être vivants, obligeant les systèmes biologiques à en supporter les conséquences tout en décodant l'information génétique de manière fiable et efficace. Des analyses s'apparentant à l'optimisation débit-distorsion en compression de données avec pertes[38] suggèrent que le code génétique résulterait de l'optimisation entre trois principes antagonistes[39] : le besoin d'un éventail d'acides aminés suffisamment diversifié[40], la nécessité de limiter l'impact des erreurs[35] et l'avantage de réduire le coût du processus en termes d'utilisation des ressources de la cellule.
Par ailleurs, la distribution des codons assignés aux acides aminés n'est pas faite au hasard[41]. Cela s'observe par le regroupement des acides aminés en codons adjacents. De plus, les acides aminés qui partagent une voie métabolique de biosynthèse commune tendent également à avoir la même première base nucléique dans leurs codons[42], tandis que ceux dont la chaîne latérale présente des propriétés physicochimiques semblables tendent à avoir également des codons semblables[43],[44], ce qui a pour effet de limiter les conséquences des mutations ponctuelles et des erreurs de traduction[41]. Enfin, une théorie permettant d'expliquer l'origine du code génétique devrait également rendre compte des observations suivantes[45] :
- l'absence de codon pour les acides aminés D ;
- le fait que seule la troisième base des codons est différente entre codons synonymes ;
- la présence de jeux de codons secondaires pour certains acides aminés ;
- la limitation à 20 acides aminés standard plutôt qu'à un nombre plus proche de 64 ;
- la relation entre les caractéristiques des codons-stop et celles des codons d'acides aminés.
Notes et références
- (en) Anton A. Turanov, Alexey V. Lobanov, Dmitri E. Fomenko, Hilary G. Morrison, Mitchell L. Sogin, Lawrence A. Klobutcher, Dolph L. Hatfield et Vadim N. Gladyshev, « Genetic Code Supports Targeted Insertion of Two Amino Acids by One Codon », Science, vol. 323, no 5911, , p. 259-261 (PMID 19131629, PMCID 3088105, DOI 10.1126/science.1164748, lire en ligne)
- (en) Christian Touriol, Stéphanie Bornes, Sophie Bonnal, Sylvie Audigier, Hervé Prats, Anne-Catherine Prats et Stéphan Vagner, « Generation of protein isoform diversity by alternative initiation of translation at non-AUG codons », Biology of the Cell, vol. 95, nos 3-4, , p. 169-178 (PMID 12867081, DOI 10.1016/S0248-4900(03)00033-9, lire en ligne)
- Ces noms de couleurs associées à des substances minérales avait été attribués par Richard Epstein et Charles Steinberg, découvreurs du codon-stop UAG, en référence à leur collègue Harris Bernstein, dont le nom de famille signifie précisément ambre en allemand : (en) Bob Edgar, « The Genome of Bacteriophage T4: An Archeological Dig », Genetics, vol. 168, no 2, , p. 575-582 (PMID 15514035, PMCID 1448817, lire en ligne)
- (en) Mario R. Capecchi, « Polypeptide Chain Termination in vitro: Isolation of a Release Factor », Proceedings of the National Academy of Sciences of the United States of America, vol. 58, no 3, , p. 1144-1151 (PMID 5233840, PMCID 335760, DOI 10.1073/pnas.58.3.1144, JSTOR 58091, Bibcode 1967PNAS...58.1144C, lire en ligne)
- (en) Eva Freisinger, Arthur P Grollman, Holly Miller et Caroline Kisker, « Lesion (in)tolerance reveals insights into DNA replication fidelity », The EMBO Journal, vol. 23, no 7, , p. 1411-1680 (PMID 15057282, PMCID 391067, DOI 10.1038/sj.emboj.7600158, lire en ligne)
- (en) J. C. Chang et Y. W. Kan, « beta 0 thalassemia, a nonsense mutation in man », Proceedings of the National Academy of Sciences of the United States of America, vol. 76, no 6, , p. 2886-2889 (PMID 88735, PMCID 383714, DOI 10.1073/pnas.76.6.2886, lire en ligne)
- (en) Séverine Boillée, Christine Vande Velde et Don W. Cleveland, « ALS: A Disease of Motor Neurons and Their Nonneuronal Neighbors », Neuron, vol. 52, no 1, , p. 39-59 (PMID 17015226, DOI 10.1016/j.neuron.2006.09.018, lire en ligne)
- (en) Dirk Isbrandt, John J. Hopwood, Kurt von Figura etChristoph Peters, « Two novel frameshift mutations causing premature stop codons in a patient with the severe form of Maroteaux-Lamy syndrome », Human Mutation, vol. 7, no 4, , p. 361-363 (PMID 8723688, DOI 10.1002/%28SICI%291098-1004%281996%297:4%3C361::AID-HUMU12%3E3.0.CO;2-0, lire en ligne)
- (en) James F. Crow, « How much do we know about spontaneous human mutation rates? », Environmental and Molecular Mutagenesis, vol. 21, no 2, , p. 122-129 (PMID 8444142, DOI 10.1002/em.2850210205, lire en ligne)
- (en) « Prevalence of positive selection among nearly neutral amino acid replacements in Drosophila », Proceedings of the National Academy of Sciences of the United States of America, vol. 104, no 16, , p. 6504-6510 (PMID 17409186, PMCID 1871816, DOI 10.1073/pnas.0701572104, Bibcode 2007PNAS..104.6504S, lire en ligne)
- (en) John W. Drake et John J. Holland, « Mutation rates among RNA viruses », Proceedings of the national Academy of Sciences of the United States of America, vol. 96, no 24, , p. 13910-13913 (PMID 10570172, PMCID 24164, DOI 10.1073/pnas.96.24.13910, lire en ligne)
- (en) J. Holland, K. Spindler, F. Horodyski, E. Grabau, S. Nichol et S. VandePol, « Rapid evolution of RNA genomes », Science, vol. 215, no 4540, , p. 1577-1585 (PMID 7041255, DOI 10.1126/science.7041255, Bibcode 1982Sci...215.1577H, lire en ligne)
- (en) J. Arjan G. M. de Visser et Daniel E. Rozen, « Clonal Interference and the Periodic Selection of New Beneficial Mutations in Escherichia coli », Genetics, vol. 172, no 4, , p. 2093-2100 (PMID 16489229, PMCID 1456385, DOI 10.1534/genetics.105.052373, lire en ligne)
- « Evidence for Stabilizing Selection on Codon Usage in Chromosomal Rearrangements of Drosophila pseudoobscura »(en)
- « Comprehensive Analysis and Comparison on the Codon Usage Pattern of Whole Mycobacterium tuberculosis Coding Genome from Different Area »(en)
- (en) Andrzej (Anjay) Elzanowski et Jim Ostell, « The Genetic Codes », sur National Center for Biotechnology Information (NCBI), (consulté le )
- (en) Nomenclature Committee of the International Union of Biochemistry (NC-IUB), « Nomenclature for Incompletely Specified Bases in Nucleic Acid Sequences », sur IUBMB, (consulté le )
- (en) T. H. Jukes et S. Osawa, « The genetic code in mitochondria and chloroplasts », Experientia, vol. 46, nos 11-12, , p. 1117-1126 (PMID 2253709, DOI 10.1007/BF01936921, lire en ligne)
- (en) David A. Fitzpatrick, Mary E. Logue, Jason E. Stajich et Geraldine Butler, « A fungal phylogeny based on 42 complete genomes derived from supertree and combined gene analysis », BMC Evolutionary Biology, vol. 6, , p. 99 (PMID 17121679, PMCID 1679813, DOI 10.1186/1471-2148-6-99, lire en ligne)
- (en) Manuel A. S. Santos et Mick F. Tuite, « The CUG codon is decoded in vivo as serine and not leucine in Candida albicans », Nucleic Acids Research, vol. 23, no 9, , p. 1481-1486 (PMID 7784200, PMCID 306886, DOI 10.1093/nar/23.9.1481, lire en ligne)
- (en) Geraldine Butler, Matthew D. Rasmussen, Michael F. Lin, Manuel A. S. Santos, Sharadha Sakthikumar, Carol A. Munro, Esther Rheinbay, Manfred Grabherr, Anja Forche, Jennifer L. Reedy, Ino Agrafioti, Martha B. Arnaud, Steven Bates, Alistair J. P. Brown, Sascha Brunke, Maria C. Costanzo, David A. Fitzpatrick, Piet W. J. de Groot, David Harris, Lois L. Hoyer, Bernhard Hube, Frans M. Klis, Chinnappa Kodira, Nicola Lennard, Mary E. Logue, Ronny Martin, Aaron M. Neiman, Elissavet Nikolaou, Michael A. Quail, Janet Quinn, Maria C. Santos, Florian F. Schmitzberger, Gavin Sherlock, Prachi Shah, Kevin A. T. Silverstein, Marek S. Skrzypek, David Soll, Rodney Staggs, Ian Stansfield, Michael P. H. Stumpf, Peter E. Sudbery, Thyagarajan Srikantha, Qiandong Zeng, Judith Berman, Matthew Berriman, Joseph Heitman, Neil A. R. Gow, Michael C. Lorenz, Bruce W. Birren, Manolis Kellis et Christina A. Cuomo, « Evolution of pathogenicity and sexual reproduction in eight Candida genomes », Nature, vol. 459, no 7247, , p. 657-662 (PMID 19465905, PMCID 2834264, DOI 10.1038/nature08064, lire en ligne)
- (en) Derek J. Taylor, Matthew J. Ballinger, Shaun M. Bowman et Jeremy A. Bruenn, « Virus-host co-evolution under a modified nuclear genetic code », PeerJ, vol. 1, , e50 (PMID 23638388, PMCID 3628385, DOI 10.7717/peerj.50, lire en ligne)
- (en) Joseph A. Krzycki, « The direct genetic encoding of pyrrolysine », Current Opinion in Microbiology, vol. 8, no 6, , p. 706-712 (PMID 16256420, DOI 10.1016/j.mib.2005.10.009, lire en ligne)
- (en) Yan Zhang, Pavel V. Baranov, John F. Atkins et Vadim N. Gladyshev, « Pyrrolysine and Selenocysteine Use Dissimilar Decoding Strategies », Journal of Biological Chemistry, vol. 280, no 21, , p. 20740-20751 (PMID 15788401, DOI 10.1074/jbc.M501458200, lire en ligne)
- (en) Laure Prat, Ilka U. Heinemann, Hans R. Aerni, Jesse Rinehart, Patrick O’Donoghue et Dieter Söll, « Carbon source-dependent expansion of the genetic code in bacteria », Proceedings of the National Academy of Sciences of the United States of America, vol. 109, no 51, , p. 21070–21075 (PMID 23185002, PMCID 3529041, DOI 10.1073/pnas.1218613110, lire en ligne)
- (en) M. Yarus, Life from an RNA World : The Ancestor Within, Cambridge, États-Unis, Harvard University Press, , 198 p. (ISBN 978-0-674-05075-4 et 0-674-05075-4, lire en ligne), p. 163
- (en) Robin D. Knight, Stephen J. Freeland et Laura F. Landweber, « Selection, history and chemistry: the three faces of the genetic code », Trends in Biochemical Sciences, vol. 24, no 6, , p. 241-247 (PMID 10366854, DOI 10.1016/S0968-0004(99)01392-4, lire en ligne)
- (en) Robin D. Knight et Laura F. Landweber, « Rhyme or reason: RNA-arginine interactions and the genetic code », Chemistry & Biology, vol. 5, no 9, , R215-R220 (PMID 9751648, DOI 10.1016/S1074-5521(98)90001-1, lire en ligne)
- (en) M. Yarus, Life from an RNA World : The Ancestor Within, Cambridge, États-Unis, Harvard University Press, , 198 p. (ISBN 978-0-674-05075-4 et 0-674-05075-4, lire en ligne), p. 170
- (en) Michael Yarus, Jeremy Joseph Widmann et Rob Knight, « RNA–Amino Acid Binding: A Stereochemical Era for the Genetic Code », Journal of Molecular Evolution, vol. 69, no 5, , p. 406-429 (PMID 19795157, DOI 10.1007/s00239-009-9270-1, lire en ligne)
- (en) Lluís Ribas de Pouplana, Robert J. Turner, Brian A. Steer et Paul Schimmel, « Genetic code origins: tRNAs older than their synthetases? », Proceedings of the National Academy of Sciences of the United States of America, vol. 95, no 19, , p. 11295-11300 (PMID 9736730, PMCID 21636, DOI 10.1073/pnas.95.19.11295, lire en ligne)
- (en) Dawn J. Brooks, Jacques R. Fresco, Arthur M. Lesk et Mona Singh, « Evolution of Amino Acid Frequencies in Proteins Over Deep Time: Inferred Order of Introduction of Amino Acids into the Genetic Code », Molecular Biology and Evolution, vol. 19, no 10, , p. 1645-1655 (PMID 12270892, DOI 10.1093/oxfordjournals.molbev.a003988, lire en ligne)
- (en) Ramin Amirnovin, « An Analysis of the Metabolic Theory of the Origin of the Genetic Code », Journal of Molecular Evolution, vol. 44, no 5, , p. 473-476 (PMID 9115171, DOI 10.1007/PL00006170, lire en ligne)
- (en) Terres A. Ronneberg, Laura F. Landweber et Stephen J. Freeland, « Testing a biosynthetic theory of the genetic code: Fact or artifact? », Proceedings of the National Academy of Sciences of the United States of America, vol. 97, no 25, , p. 13690-13695 (PMID 11087835, PMCID 17637, DOI 10.1073/pnas.250403097, lire en ligne)
- (en) Stephen J. Freeland, Tao Wu et Nick Keulmann, « The Case for an Error Minimizing Standard Genetic Code », Origins of life and evolution of the biosphere, vol. 33, nos 4-5, , p. 457-477 (PMID 14604186, DOI 10.1023/A:1025771327614, lire en ligne)
- (en) Pavel V. Baranov, Maxime Venin et Gregory Provan, « Codon Size Reduction as the Origin of the Triplet Genetic Code », PLoS One, vol. 4, no 5, , e5708 (PMID 19479032, PMCID 2682656, DOI 10.1371/journal.pone.0005708, lire en ligne)
- (en) Tsvi Tlusty, « A model for the emergence of the genetic code as a transition in a noisy information channel », Journal of Theoretical Biology, vol. 249, no 2, , p. 331-342 (PMID 17826800, DOI 10.1016/j.jtbi.2007.07.029, lire en ligne)
- (en) Tsvi Tlusty, « Rate-Distortion Scenario for the Emergence and Evolution of Noisy Molecular Codes », Physical Reviews Letters, vol. 100, no 4, , p. 048101 (PMID 18352335, DOI https://dx.doi.org/10.1103/PhysRevLett.100.048101, lire en ligne)
- (en) Tsvi Tlusty, « A colorful origin for the genetic code: Information theory, statistical mechanics and the emergence of molecular codes », Physics of Life Reviews, vol. 7, no 3, , p. 362-376 (PMID 20558115, DOI 10.1016/j.plrev.2010.06.002, Bibcode 2010PhLRv...7..362T, lire en ligne)
- (en) Guy Sella et David H. Ardell, « The Coevolution of Genes and Genetic Codes: Crick’s Frozen Accident Revisited », Journal of Molecular Evolution, vol. 63, no 3, , p. 297-313 (PMID 16838217, DOI 10.1007/s00239-004-0176-7, lire en ligne)
- (en) Stephen J. Freeland et Laurence D. Hurst, « The Genetic Code Is One in a Million », Journal of Molecular Evolution, vol. 47, no 3, , p. 238-248 (PMID 9732450, DOI 10.1007/PL00006381, lire en ligne)
- (en) F. J. R. Taylor et D. Coates, « The code within the codons », Biosystems, vol. 22, no 3, , p. 177-187 (PMID 2650752, DOI 10.1016/0303-2647(89)90059-2, lire en ligne)
- (en) Massimo Di Giulio, « The extension reached by the minimization of the polarity distances during the evolution of the genetic code », Journal of Molecular Evolution, vol. 29, no 4, , p. 288-293 (PMID 2514270, DOI 10.1007/BF02103616, lire en ligne)
- (en) J. T. Wong, « Role of minimization of chemical distances between amino acids in the evolution of the genetic code », Proceedings of the National Academy of Sciences of the United States of America, vol. 77, no 2, , p. 1083-1086 (PMID 6928661, PMCID 348428, DOI 10.1073/pnas.77.2.1083, Bibcode 1980PNAS...77.1083W, lire en ligne)
- (en) Albert Erives, « A Model of Proto-Anti-Codon RNA Enzymes Requiring l -Amino Acid Homochirality », Journal of Molecular Evolution, vol. 73, nos 1-2, , p. 10-22 (PMID 21779963, PMCID 3223571, DOI 10.1007/s00239-011-9453-4, lire en ligne)
Voir aussi
Article connexe
Lien externe
- Portail de la biologie cellulaire et moléculaire
- Portail de la médecine