Chaîne la plus proche
En informatique théorique, et notamment en algorithmique du texte, la chaîne la plus proche (en anglais closest string) d'un ensemble de chaînes de caractères données est une chaîne à distance minimale des chaînes, selon la distance de Hamming. La recherche de la chaîne la plus proche est un problème algorithmique NP-difficile[1].
Exemple
Pour les cinq chaînes
BARACADABRA ABRAACDABRA RBRACADABCA ACRACADABCA ABRADADABCA
la chaîne la plus proche est
ABRACADABRA
Elle est à distance 2 des chaînes données. Les coïncidences sont marquées en rouge :
BARACADABRA ABRAACDABRA RBRACADABCC ACRACADABCA ABRADADABCA
Définition formelle
Étant donné n chaînes s1, ..., sn de même longueur m, le problème de la chaîne la plus proche consiste à déterminer une nouvelle chaîne s de longueur m telle que d(s,si) ≤ k pour tout i, où d est la distance de Hamming, et où k est aussi petit que possible[2]. Le problème de décision correspondant, qui est NP-complet, prend également l'entier k en entrée et demande s'il existe une chaîne qui est à distance de Hamming au plus k de toutes les chaînes données[1]. Le problème est NP-difficile même sur un alphabet binaire[3].
Motivation
En bio-informatique, le problème de la chaîne la plus proche est un aspect très étudié du problème de détection de signaux dans les ADN. Le problème a aussi des applications en théorie des codes (problème du rayon minimal)[3]. Le problème de la sous-chaîne la plus proche formalise des applications biologiques dans la recherche de régions conservées, l'identification de cibles de médicaments génétiques et la génération de sondes génétiques en biologie moléculaire. En algorithmique du texte, c'est un problème de combinatoire de chaînes de caractères[4]. Ce problème de chaînes et d'autres qui se posent dans diverses applications allant de l'exploration de texte à l'analyse de séquence biologique sont souvent NP-difficiles. Ceci motive la recherche de cas spéciaux traitables (fixed-parameter tractable) de ces problèmes. Ceci a un sens dans les cas où il y a plusieurs paramètres naturellement associés, comme la taille de l’alphabet, le nombre de chaînes, ou un majorant sur la distance.
Le problème de la chaîne la plus proche peut être vu géométriquement comme une instance du problème du 1-centre avec comme distance la distance de Hamming.
Simplifications et réduction de données
Des instances du problème de la chaîne la plus proche peuvent contenir des informations qui ne sont pas significatives pour le problème ou, autrement dit, qui ne contribuent pas à la difficulté du problème. Par exemple, si certaines chaînes contiennent le caractère a, et qu'aucune ne contient le caractère z, remplacer tous les a par des z donne une instance essentiellement équivalente, c'est-à-dire qu'à partir d'une solution de l'instance modifiée, la solution originale peut être facilement restaurée et vice-versa.
Normalisation de l'entrée
En écrivant les chaînes d'entrée les unes sous les autres, on obtient une matrice. Certains types de lignes ont essentiellement les mêmes influences sur la solution. Par exemple, remplacer une colonne qui a les entrées (a, a, b) par une colonne (x, x, y) peut mener à une chaîne solution différente, mais ne peut affecter la solvabilité, car les deux colonnes expriment la même structure, où les deux premières entrées sont égales, mais différentes de la troisième.
Une instance d'entrée peut être normalisée en remplaçant, dans chaque colonne, le caractère qui se produit le plus souvent par a, le caractère qui se produit en deuxième par b, et ainsi de suite. Étant donné une solution de l'instance normalisée, l'instance d'origine peut être retrouvée en remplaçant les caractères de la solution par leur version d'origine dans chaque colonne. L'ordre des colonnes ne contribue pas à la difficulté du problème. Cela signifie que si on permute les chaînes d'entrée selon une certaine permutation π et si on obtient une chaîne solution s à cette instance modifiée, alors π−1(s) est une solution de l'instance d'origine.
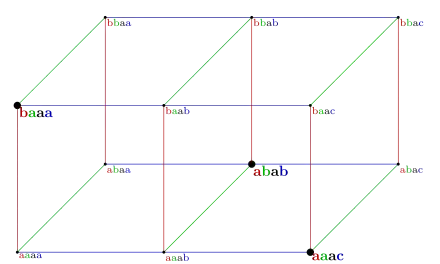
Exemple
On considère une instance formée des trois mots uvwx, xuwv, et xvwu (voir schéma en haut). Sous forme de matrice, on obtient la table suivante :
u v w x x u w v x v w u
Dans la première colonne (u, x, x), c'est le symbole x qui apparaît le plus souvent (2 fois) ; on le remplace par a et le caractère u par b, ce qui donne la nouvelle première colonne (b, a, a). La deuxième colonne (v, u, v) est remplacée, par la même stratégie, par (a, b, a). En procédant de même pour les autres colonnes, on aboutit à
b a a a a b a b a a a c
Réduction de données par normalisation
La normalisation a pour conséquence que la taille de l'alphabet est au plus égale au nombre de chaînes d'entrées. Ceci peut s'avérer utile pour des algorithmes dont le temps de calcul dépend de la taille de l’alphabet.
Approximation
Li et al. ont décrit un schéma d'approximation en temps polynomial[5], mais qui est inutilisable en pratique à cause de grandes constantes cachées.
Complexité paramétrée
Le problème de la chaîne la plus proche peut être résolu en O(nL+nd × dd), où n est le nombre de chaînes données en entrée, L est la longueur commune de toutes les chaînes et d est la distance désirée de la solution à toutes les chaînes données[6]. Il est ainsi dans la classe de complexité paramétrée FPT (fixed-parameter tractable).
Relations avec d'autres problèmes
Le problème de la chaîne la plus proche est un cas particulier du problème plus général de la sous-chaine la plus proche (en) qui est strictement plus difficile . Alors que le problème de la chaîne la plus proche est fixed parameter tractable de différentes manières, le problème de la sous-chaine W[1]-difficile par rapport à ces paramètres. La kernelisation de ces problèmes et de problème voisin a fait l'objet d'une étude détaillée qui montre que soit on peut obtenir un noyau polynomial soit qu'un tel noyau ne peut être obtenu sous les hypothèses usuelles en théorie de la complexité, et notamment la Exponential Time Hypothesis (ETH)[7]. Ces problèmes sont de trouver une sous-structure ou une superstructure commune à ensemble de chaînes donné. Les problèmes les plus fondamentaux sont la plus longue sous-séquence commune, la plus courte super-séquence commune et la plus petite séquence commune. En outre, il y a le problème plus général de l'alignement de séquences multiples, qui est d'une importance capitale dans l'analyse des séquences biologiques[4].
Notes et références
- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « closest string » (voir la liste des auteurs).
- J. Kevin Lanctot, Ming Li, Bin Ma, Shaojiu Wang et Louxin Zhang, « Distinguishing string selection problems », Information and Computation, vol. 185, no 1, , p. 41–55 (DOI 10.1016/S0890-5401(03)00057-9, Math Reviews 1994748)
- Bin Ma et Xiaming Sun, « More Efficient Algorithms for Closest String and Substring Problems », dans Proc. 12th Ann. Int. Conf. on Research in Computational Molecular Biology (RECOMB), Springer, coll. « Lecture Notes in Computer Science » (no 4955), , 396-409 p. (DOI 10.1007/978-3-540-78839-3_33, lire en ligne).
- Moti Frances et Ami Litman, « On covering problems of codes », Theory of Computing Systems, vol. 30, no 2, , p. 113-119.
- Laurent Bulteau, Falk Hüffner, Christian Komusiewicz et Rolf Niedermeier, « Multivariate algorithmics for NP-hard string problems », Bulletin of the EATCS, vol. 114, (lire en ligne, consulté le ).
- Ming Li, Bin Ma et Lusheng Wang., « On the Closest String and Substring Problems. », Journal of the ACM, vol. 49, no 2, , p. 157-171 (DOI 10.1145/506147.506150, lire en ligne)
- Jens Gramm, Rolf Niedermeier et Peter Rossmanith, « Fixed-Parameter Algorithms for Closest String and Related Problems », Algorithmica, vol. 37, , p. 25–42 (DOI 10.1007/s00453-003-1028-3, lire en ligne)
- Manu Basavaraju, Fahad Panolan, Ashutosh Rai, M.S. Ramanujan et Saket Saurabh, « On the kernelization complexity of string problems », Theoretical Computer Science, vol. 730, , p. 21-31 (DOI 10.1016/j.tcs.2018.03.024).
Articles liés
- Algorithmique du texte
- Séquence biologique
- Alignement de séquences
- Exponential time hypothesis (en)
- Complexité paramétrée
- Kernelisation
- Portail de l'informatique théorique