Acide ribonucléique messager
L'acide ribonucléique messager, ARN messager, ou ARNm (en anglais, mRNA, pour messenger ribonucleic acid), est une copie transitoire d'une portion de l'ADN correspondant à un ou plusieurs gènes. L'ARNm est utilisé comme intermédiaire par les cellules pour la synthèse des protéines. Le concept d'ARN messager a été émis puis démontré par Jacques Monod, François Jacob et leurs collaborateurs à l'Institut Pasteur en 1961[1], ce qui leur a valu le prix Nobel en 1965[2],[3].
Pour les articles homonymes, voir messager.
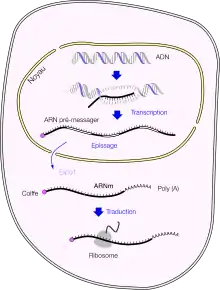
L'ARNm est une copie simple brin linéaire de l'ADN composée d'ARN, qui comprend la région codant une protéine encadrée de régions non codantes. Il est synthétisé sous forme de précurseur dans le noyau de la cellule lors d'un processus appelé transcription. Il subit alors plusieurs étapes de maturation, ses deux extrémités sont modifiées, certaines régions non codantes appelées introns peuvent être excisées lors d'un processus appelé épissage. L'ARNm maturé est exporté dans le cytoplasme où il est traduit en protéine par un ribosome. L'information portée par l'ARNm est constituée d'une série de codons, des triplets de nucléotides consécutifs qui codent chacun un acide aminé de la protéine correspondante. L'enchaînement de ces codons constitue le gène proprement dit ou cistron. La correspondance entre codons et acides aminés constitue ce qu'on appelle le code génétique.
La transcription des ARNm et leur traduction sont des processus qui sont l'objet de contrôles cellulaires importants et permettent à la cellule de réguler l'expression des différentes protéines dont elle a besoin pour son métabolisme.
Chez les eucaryotes, un ARNm correspond en général à un seul gène et code une seule protéine (un seul cadre de lecture ouvert). Ce sont des ARN monocistroniques. Chez les bactéries, les ARNm peuvent en revanche coder plusieurs protéines (plusieurs phases ouvertes de lectures successives): on les qualifie alors d'ARN polycistroniques. Chez ces organismes qui ne possèdent pas de noyau, la transcription des ARN messagers et leur traduction en protéine sont le plus souvent couplées.
Rôle cellulaire
L'information génétique d'une cellule qui gouverne l'essentiel des aspects de son fonctionnement est contenue dans son génome qui est constitué d'ADN. Cette information doit être traduite sous la forme de protéines, qui sont les molécules effectrices de l'organisme vivant. Plutôt que d'utiliser directement la matrice d'ADN pour synthétiser des protéines, l'évolution a établi une molécule intermédiaire, l'ARN messager[3]. Cet ARNm est une copie transitoire de la partie de l'ADN contenant les instructions d'assemblage d'une protéine, c'est-à-dire son gène. En effet, à un gène donné correspond en général une protéine.
La présence des ARNm en tant que molécules intermédiaires permet aussi de réguler l'expression des gènes. Les besoins cellulaires pour une protéine donnée peuvent varier en fonction des conditions environnementales, du type cellulaire, du stade de développement, de l'âge de la cellule. Les ARNm sont des molécules labiles, dont la durée de vie est limitée, variant de quelques minutes à quelques heures. Leur production peut être adaptée par la cellule aux conditions spécifiques auxquelles la cellule est confrontée. Lorsqu'une protéine est nécessaire, la cellule transcrit l'ARNm correspondant. Lorsqu'à l'inverse elle n'en a plus besoin, la transcription du gène s'arrête et l'ARNm est progressivement dégradé par des ribonucléases (ou RNases). Ainsi la production de protéine peut être stimulée ou réprimée en fonction des besoins.
La régulation de la production d'une protéine à partir de son gène peut s'effectuer à plusieurs niveaux : par la régulation de la transcription de l'ADN en ARNm que l'on appelle le contrôle transcriptionnel, ou par le contrôle de la traduction de l'ARNm en protéine, que l'on appelle le contrôle traductionnel ou régulation de la traduction. La cellule peut donc « choisir » quelles parties de l'ADN seront transcrites et ainsi exprimées. Les différentes cellules expriment différentes parties du génome pour obtenir un phénotype différent selon la présence de facteurs de régulation.
Structure
Comme tous les ARN, l'ARNm est un acide nucléique résultant de la polymérisation de ribonucléotides reliés par des liaisons phosphodiester. Comme le terme acide ribonucléique l'indique, l'ose (ou plus familièrement « sucre ») présent dans les ribonucléotides est le ribose. Les bases nucléiques, ou bases azotées, présentes sur les ribonucléotides sont l'adénine (A) qui est complémentaire de l'uracile (U) et la guanine (G) complémentaire de la cytosine (C). L'ARNm emploie donc la base U, à la différence de l'ADN qui utilise la base T (la thymine)[4]. Contrairement à l'ADN, l'ARNm est une molécule monocaténaire, c’est-à-dire formée d'un seul brin. On distingue trois principales régions fonctionnelles dans un ARNm : la région 5′ non traduite (5'-UTR), le ou les cistrons codants et enfin la région 3′ non traduite (3'-UTR). Les deux régions non traduites ou régions UTR (de l'anglais untranslated regions) contiennent souvent des signaux d'expression ou de maturation de l'ARN.

Ce caractère simple brin de l'ARNm n'empêche pas des repliements locaux complexes et parfois très structurés de la molécule sur elle-même faisant intervenir la complémentarité entre bases nucléiques. On distingue deux niveaux d'organisation structurale de la molécule d'ARN : la structure secondaire, qui est définie par les appariements dits Watson-Crick entre bases, et la structure tertiaire, qui est une étape supplémentaire de repliement en trois dimensions de la molécule.
Dans le cas des ARNm, les structures secondaires peuvent jouer un rôle dans la régulation de l'expression des gènes, en modulant l'efficacité d'une ou plusieurs des étapes de transcription et de traduction. Par exemple, la présence de structures secondaires de la région 5′ non traduite peut influencer le recrutement du ribosome et donc l'efficacité de la traduction. Les structures secondaires peuvent également être des sites de liaisons pour des protéines qui modulent l'épissage ou la polyadénylation.
Synthèse et maturation
Transcription
L'ARNm est une copie d'une région de l'ADN correspondant à un ou quelques gènes codant des protéines. L'opération de copie, appelée transcription, se déroule dans le noyau de la cellule chez les eucaryotes et dans le cytoplasme chez les procaryotes, c'est-à-dire là où réside le matériel génétique. La transcription a aussi lieu dans les organites semi-autonomes comme les mitochondries et les chloroplastes qui possèdent un génome indépendant. Cette transcription est effectuée par des enzymes spécifiques appelées ARN polymérases qui utilisent des ribonucléotides triphosphates comme monomères pour la synthèse de l'ARN. Chez les eucaryotes qui possèdent trois ARN polymérases distinctes dans leur noyau, c'est l'ARN polymérase II qui transcrit tous les ARNm cytoplasmiques, les deux autres ARN polymérases assurant la synthèse d'ARN stables non codants (ARN ribosomique, ARN de transfert…).
L'ARN polymérase se fixe sur une séquence spécifique de l'ADN appelée promoteur de transcription, juste en amont du début de l'ARNm. Elle sépare alors les deux brins du duplex d'ADN et crée ce qu'on appelle une bulle de transcription puis synthétise la molécule d'ARN en se servant du brin d'ADN dit brin transcrit comme matrice. Un signal de terminaison, situé en aval du ou des gènes transcrits déclenche l'arrêt de la transcription, le détachement de l'ARN polymérase et la libération de l'ARNm terminé.
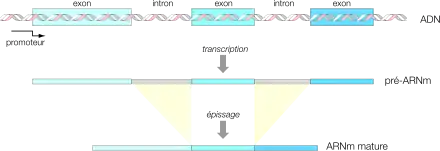
Chez les procaryotes, la molécule d'ARNm est le plus souvent directement traduite en protéines. Chez les eucaryotes (noyau et organites), la molécule synthétisée par les ARN polymérases est dans de nombreux cas de l'ARN pré-messager qui doit subir des maturations post-transcriptionnelles dans le noyau avant de pouvoir être traduit. En particulier, les parties codantes chez les eucaryotes sont en général interrompues par des séquences non codantes ou introns qui séparent les exons codants. Au cours de la maturation post-transcriptionnelle aura notamment lieu le phénomène d'épissage assurant la suture des séquences résultant de la transcription des exons, alors que les séquences résultant de la transcription des introns seront retirées de l'ARN pré-messager.
Modifications post-transcriptionnelles
Chez les eucaryotes, après transcription de l'ADN, on obtient une molécule d'ARN précurseur ou ARN pré-messager qui n'est pas encore « mature », et qui ne peut pas, dans la majorité des cas, être directement traduite en protéine. Pour transformer ce pré-ARNm en ARNm mature, compétent pour la traduction, il faut lui faire subir deux types de modifications. Il faut modifier ses extrémités 5′ (la coiffe) et 3′ (queue poly(A)) afin en particulier d'augmenter sa stabilité mais aussi d'en permettre l'adressage vers la machinerie cellulaire adéquate, c'est-à-dire le ribosome dans le cytoplasme. Il faut aussi réaliser une étape complexe appelée épissage. Le pré-ARNm contient en effet dans sa structure une alternance d'introns et d'exons, dont seuls les exons codent la synthèse de la protéine. Les introns doivent donc être épissés (retirés) avant que le processus de traduction puisse s'effectuer. À l'issue de ces étapes qui se déroulent dans le noyau, on obtient un ARNm qui est alors exporté dans le cytoplasme.
Plusieurs facteurs interviennent dans ce processus. Après la transcription, le pré-ARNm s'associe à des protéines nucléaires pour former des ribonucléoprotéines nucléaires hétérogènes (hnRNP) qui interviennent dans la maturation et l'export. Plusieurs enzymes de modification du pré-ARNm sont directement associés à l'ARN polymérase II, ce qui permet d'agir sur le pré-ARNm au cours même de sa synthèse. On parle alors de modifications co-transcriptionnelles. Enfin, le processus d'épissage (voir plus bas) fait intervenir des petits ARN nucléaires (ARNsn) au sein de complexes snRNP. Ces complexes s'associent et se dissocient de manière ordonnée aux jonctions intron-exon pour en réaliser la coupure et la suture.
Modifications en 5′
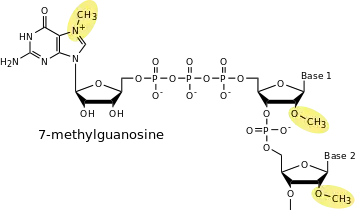
Chez les eucaryotes, l'extrémité 5′ de l'ARNm subit une série d'étapes post-transcriptionnelles qui vont aboutir à la formation de ce qu'on appelle la coiffe[5]. Le rôle de cette coiffe est multiple. Elle permet de stabiliser l'ARNm en le protégeant contre l'action de ribonucléases. La coiffe intervient également dans l'export de l'ARNm dans le cytoplasme (voir plus bas) et dans le recrutement du ribosome pour la traduction.
La formation de la coiffe est co-transcriptionnelle, elle se produit très rapidement après le début de la synthèse par l'ARN polymérase II, lorsque l'ARNm ne comporte qu'environ une trentaine de nucléotides. Ceci résulte du fait que ces modifications sont catalysées par des enzymes qui sont associées à l'ARN polymérase. Le synoptique des évènements est le suivant.
La première étape est le clivage de l'extrémité 5′-triphosphate de l'ARN précurseur par une triphosphatase. La triphosphatase laisse une extrémité 5′-diphosphate. Il y a ensuite ajout d'une guanosine triphosphate par une guanylyl-transférase. Cette enzyme forme un pont 5′-5′ triphosphate dans lequel la guanosine additionnelle est orientée tête-bêche. Il y a ensuite une série de méthylations, sur la position N7 de la guanosine ajoutée et sur les 2′-hydroxyles des deux premiers riboses. Enfin, lorsque le premier nucléotide transcrit est une adénosine, on peut avoir la formation de N6-méthyl-adénosine, par l'action d'une ARN méthylase qui est toutefois différente de l'ADN méthylase de maintenance.
L'ensemble de ces modifications a pour but de faire barrière à l'action des 5′-exonucléases, donc de protéger l'ARN de son environnement, mais joue aussi un rôle dans l'épissage, dans le transport nucléo-cytoplasmique et dans la traduction de l'ARNm en protéines.
Terminaison et modification en 3′
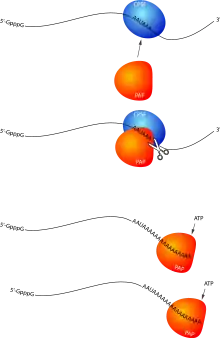
Chez les eucaryotes, la terminaison de la transcription se produit lorsque l'ARN polymérase atteint le signal de polyadénylation, 5′-AAUAAA-3′, dans la région 3′-UTR de l'ARNm. Il y a alors recrutement d'un complexe protéique de coupure, CPSF (cleavage and polyadenylation specificity factor). Le rôle de ce facteur est double : en interagissant avec l'ARN polymérase, il va provoquer l'arrêt de la transcription et le détachement de cette dernière ; ensuite il va cliver l'ARN juste en aval du signal AAUAAA, au niveau d'une séquence de type 5′-YA-3′, où Y est une base pyrimidique (U ou C).
Après la coupure, CPSF va recruter la poly(A) polymérase qui va synthétiser une queue poly(A) composée de 100 à 250 résidus adényliques[6]. Cette queue poly(A) joue plusieurs rôles, via le recrutement de protéines spécifiques, les PABP (poly(A) binding proteins). La polyadénylation et la fixation des PABP sur l'ARNm sont importantes pour l'export nucléaire[7], la protection contre les ribonucléases mais aussi 3′-OH, mais surtout pour la traduction en protéine (voir plus bas).
Chez les bactéries, le processus de terminaison de la transcription est différent, il passe principalement par la formation d'une structure secondaire sur l'ARNm (le terminateur), qui provoque une pause de l'ARN polymérase et son détachement de l'ADN matrice. Les ARNm bactériens ne sont en général pas polyadénylés en 3′.
Épissage
Les gènes eucaryotes ont fréquemment une structure mosaïque où les régions codant une protéine sont morcelées en plusieurs segments (exons) séparés par des régions non codantes appelées introns. La transcription du gène produit initialement un pré-ARNm dans lequel exons et introns alternent. L'épissage est le processus de maturation se déroulant dans le nucléoplasme qui permet l'excision des introns et la suture des différents exons pour obtenir un ARNm mature dans lequel le cadre de lecture de la protéine a été reconstitué.
Les jonctions amont et aval de chaque intron se caractérisent par des séquences de nucléotides conservées, qui sont reconnues par la machinerie d'épissage. Il existe également une séquence conservée à l'intérieur de l'intron, appelée boîte de branchement, qui contient une adénosine strictement conservée essentielle au mécanisme.
L'épissage est réalisé par un ensemble de cinq particules ribonucléoprotéiques, les snRNP, chacune composée d'un ARN et de plusieurs protéines. Ces cinq snRNP, qui ont pour noms U1, U2, U4, U5 et U6, forment le splicéosome. Elles interviennent en séquence et se lient au pré-ARNm par le biais d'appariements de bases de type Watson-Crick.

Mécanisme de l'épissage
La première étape de l'épissage est une attaque de la liaison phosphodiester à la jonction entre le côté 5′ de l'intron et l'exon amont. C'est le 2′-OH du ribose de l'adénosine conservée dans la boîte de branchement qui joue le rôle de nucléophile dans cette réaction qui conduit à la formation d'un intermédiaire cyclique appelé lasso (en anglais lariat) par transestérification. Dans cet intermédiaire il y a formation d'une liaison 2′-5′ phosphodiester entre la guanosine conservée à l'extrémité de l'intron et l'adénosine. Cette dernière est ainsi impliquée dans trois liaisons phosphodiester, en 5′ et 3′ avec les nucléotides précédents et suivants du pré-ARNm, et en 2′ avec l'extrémité 5′ de l'intron.
Ce premier transfert libère une extrémité 3′-OH libre sur l'exon amont. Celle-ci attaque alors à son tour la jonction aval de l'intron par un mécanisme analogue de transestérification, ce qui aboutit à la suture des deux exons. L'intron sous forme de lasso est libéré et sera ultérieurement ouvert par une enzyme de débranchement pour permettre son recyclage.
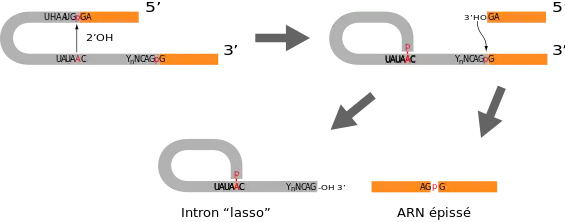
Les snRNP interviennent dans ce processus en s'appariant aux jonctions de l'intron et des exons et à la boîte de branchement. Ils maintiennent les différents acteurs pendant les deux étapes de transestérification et assurent le positionnement correct des différents groupements -OH réactifs. Le snRNP U1 se fixe initialement sur la jonction exon-intron amont et le snRNP U2 sur la boîte de branchement ; U5 maintient les deux bords des deux exons et U6 joue probablement un rôle catalytique.
Édition
L'édition est un phénomène biologique qui permet à la cellule de modifier la séquence de l'ARN messager après la transcription. La séquence polypeptidique qui résultera de la traduction de cet ARNm ne correspond donc pas à la séquence exacte du gène correspondant. C'est une forme de régulation post-transcriptionnelle, ou de maturation de l'ARN. Ce processus, découvert initialement chez les mitochondries de trypanosomes[8], implique des facteurs protéiques et parfois des ARN guides. Il conduit soit à l'incorporation de nucléotides additionnels qui sont insérés dans l'ARNm, soit à la conversion chimique de certaines bases, par exemple du fait de l'action d'enzymes comme les cytidines ou les adénosines désaminases.
Export du noyau
Lors du processus de synthèse et de maturation dans le noyau, le pré-ARNm puis l'ARNm maturé interagit avec un certain nombre de protéines nucléaires de manière séquentielle. Il forme ainsi un complexe ribonucléique appelé hnRNP (heterogeneous nuclear ribonucleoprotein). Ce complexe contient en particulier une protéine qui lie la coiffe, le CBC (cap-binding complex), une protéine qui se lie à la queue poly(A) et des protéines d'adressage au pore nucléaire, le complexe Nxt1-Nxf1. Ce complexe d'export interagit avec des protéines spécifiques du pore, les FG-Nups qui permettent ensuite la translocation de l'ARNm vers le cytoplasme.
La formation d'un complexe de l'ARNm avec les protéines d'adressage au pore, Nxt1-Nxf1, est dépendante de la maturation correcte de l'ARNm, et en particulier de la présence de la coiffe et de l'épissage. C'est une première étape de contrôle qualité : les ARNm incomplètement maturés ne sont pas exportés dans le cytoplasme.
Le mécanisme d'export des ARNm est donc spécifique et différent des autres mécanismes de transport nucléo-cytoplasmique qui utilisent des karyophérines, importines ou exportines, pour assurer le transport de cargaison au travers du pore. Il diffère donc en particulier du mécanisme d'export des ARNt qui transitent quant à eux associés à une exportine.
Transport et adressage
Dans les cellules, les ARNm peuvent être dirigés vers des compartiments particuliers pour y être stockés ou traduits dans un site donné. C'est en particulier le cas pour les neurones qui sont des cellules de grandes dimensions. Certaines protéines ne sont produites qu'au niveau de la synapse, à l'extrémité de l'axone ou des dendrites, leur ARNm est donc transporté de manière active jusqu'à celles-ci pour y être traduit[9].
Traduction en protéine
Les ARNm maturés sont ensuite traduits en protéines par les ribosomes dans le cytoplasme. La petite sous-unité du ribosome (30S chez les bactéries ou 40S chez les eucaryotes) se fixe d'abord dans la région amont de l'ARNm et glisse jusqu'au codon de démarrage. Cette étape nécessite l'intervention d'un ensemble de protéines spécifiques appelées facteurs d'initiation. Il recrute alors le premier ARN de transfert et la grande sous-unité du ribosome (50S chez les bactéries et 60S chez les eucaryotes) s'associe à l'ensemble. Le ribosome ainsi assemblé démarre la traduction[10].
À la fin de cette phase d'initiation, les facteurs d'initiation quittent le ribosome assemblé qui va allonger la protéine codée par l'ARNm, au cours de cycles d'élongations successives. À chaque cycle, le ribosome lit un codon et y associe l'ARNt de codon complémentaire, qui porte l'acide aminé correspondant. Cette étape nécessite l'action de facteurs d'élongation. Une fois l'ensemble du cistron lu par le ribosome, celui-ci parvient sur le codon d'arrêt qui le termine. Le ribosome libère la protéine terminée, puis le dernier ARNt et enfin l'ARNm. Ces étapes nécessitent l'intervention de facteurs de terminaison.
Les différentes étapes impliquées sont détaillées ci-après.
ARNm dans le cytoplasme
Dans le cytoplasme des cellules eucaryotes, les ARNm existent sous forme de complexes associés à des protéines, sous forme pseudo-circulaire. La coiffe en 5′ est en effet reconnue par le facteur de démarrage de la traduction eIF4E. La queue poly(A) est recouverte par une protéine spécifique, la PABP (poly(A) binding protein). Ces protéines, eIF4E et PABP interagissent toutes les deux avec une même protéine organisatrice, le facteur de démarrage eIF4G[11]. Ceci conduit à une circularisation de l'ARNm au travers de ce complexe protéique, ses deux extrémités 5′ et 3′ étant ainsi rapprochées. Ce complexe contient aussi d'autres facteurs de démarrage, comme eIF4A qui est une ARN hélicase.
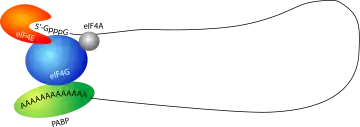
L'un des effets de la formation de ces complexes circulaires est de permettre la rapprochement de la 5'-UTR et de la 3'-UTR de l'ARNm. Ces deux régions peuvent contenir des signaux d'expression qui vont ainsi pouvoir jouer conjointement sur le recrutement du ribosome. C'est aussi un moyen pour la cellule de vérifier que l'ARNm est complet et contient bien simultanément une coiffe et une queue poly(A) avant de démarrer la traduction.
Recrutement du ribosome
Le recrutement du ribosome sur l'ARNm est une étape clé de la synthèse des protéines. L'efficacité de cette étape détermine en particulier le taux de synthèse final de la ou des protéines codées par l'ARNm. C'est à ce niveau que s'exercent la plupart des régulations dites traductionnelles de l'expression du génome (par opposition aux régulations transcriptionnelles qui affectent le taux de synthèse de l'ARNm). Le mécanisme de recrutement du ribosome est différent chez les eucaryotes et chez les bactéries. Chez ces dernières, la petite sous-unité du ribosome (30S) procède par un mécanisme d'entrée interne sur l'ARNm, directement sur le codon de démarrage. À l'inverse, chez les eucaryotes, la sous-unité 40S est recrutée à l'extrémité 5′ de l'ARNm, sur la coiffe, et procède par un mécanisme de balayage (scanning)[10]. Enfin, il existe dans certains cas un mécanisme d'entrée interne du ribosome chez les eucaryotes, en particulier chez certains ARNm viraux. Ceux-ci nécessitent des structures particulières sur l'ARNm, appelées IRES.
Démarrage de la traduction procaryote
Chez les bactéries, le démarrage de la traduction des ARNm correspond à la reconnaissance du codon de démarrage. Celui-ci est un codon AUG, GUG ou plus rarement UUG, qui doit être précédé sur l'ARNm par une séquence conservée riche en purine, appelée RBS (ribosome-binding site) ou séquence de Shine & Dalgarno[12]. Ce RBS est en général séparé du codon de démarrage par 6 à 12 nucléotides. Sa séquence est complémentaire de l'extrémité 3′ de l'ARN ribosomique 16S, il se forme ainsi un appariement entre ARNm et ARNr, juste en amont du codon de démarrage qui se trouve ainsi pré-positionné dans le site P, dans la sous-unité 30S du ribosome.
La formation du complexe de démarrage fait intervenir trois facteurs d'initiation, appelés IF1, IF2 et IF3. IF1 vient se localiser au niveau du site A de la sous-unité 30S et en bloque ainsi l'accès pendant la phase de démarrage. IF2 s'associe à l'ARNt initiateur portant une formyl-méthionine qui sera le premier acide aminé incorporé dans la protéine. IF3 s'associe à la sous-unité 30S, dont il empêche la réassociation avec la sous-unité 50S.
Le processus se déroule de la manière suivante : la sous-unité 30S associée à IF3 balaie l'ARNm jusqu'à trouver un site RBS auquel elle s'apparie. L'ARNt associé à IF2 rentre dans le site P, canalisé dans ce processus par IF1 qui bloque l'autre site. L'interaction codon-anticodon entre l'ARNm et l'ARNt cale le ribosome sur le bon cadre de lecture ouvert. IF2 hydrolyse une molécule de GTP, IF3 et IF1 quittent le complexe. La sous-unité 50S peut alors s'associer. Le ribosome complet peut alors commencer la traduction au niveau du second codon du cistron.
Démarrage de la traduction eucaryote
Chez les eucaryotes, les ARNm matures existent sous forme de complexes pseudo-circulaires dans le cytoplasme (voir plus haut), associant les facteurs de démarrage eIF4E, eIF4G et eIF4A, ainsi que la PABP. C'est le facteur eIF4G qui joue le rôle de protéine organisatrice dans ce complexe car il est lié simultanément à la coiffe via eIF4E et à la queue poly(A) via la PABP. Lorsque ces deux interactions sont réalisées, eIF4G peut effectuer le recrutement de la petite sous-unité (40S) du ribosome.
Cette dernière est elle-même associée à plusieurs facteurs au sein d'un complexe de pré-initiation 43S. Celui-ci contient, outre la sous-unité ribosomique 40S, l'ARNt de démarrage aminoacylé par la méthionine associé au facteur eIF2 complexé au GTP, le facteur eIF3 qui maintient la sous-unité 40S dissociée de la grande sous-unité, ainsi que eIF1A et eIF5. Au sein de ce complexe de pré-initiation, eIF3 interagit avec eIF4G au sein du complexe ARNm. Ceci permet le recrutement du ribosome au niveau de la coiffe de l'ARNm. Le complexe d'initiation complet ainsi formé est appelé complexe 48S[13]. Il contient donc :
- l'ARNm coiffé et polyadénylé ;
- la sous-unité 40S du ribosome ;
- l'ARNt de démarrage au site P de la précédente ; cet ARNt est aminoacylé par la méthionine et associé à eIF2:GTP ;
- les facteurs de démarrage eIF1, eIF1A, eIF2 (déjà cité), eIF3, eIF4A, eIF4B, eIF4E, eIF4G et eIF5.
Ce complexe 48S, recruté sur la coiffe de l'ARNm, va alors avancer le long de l'ARNm jusqu'à atteindre le premier triplet AUG qui va être reconnu comme codon d'initiation. Cette reconnaissance est renforcée par la présence de nucléotides conservés autour du codon AUG qui renforcent l'interaction avec le ribosome. Ce consensus est appelé séquence de Kozak[14]. Il se forme alors une interaction entre l'anticodon de l'ARNt et ce codon AUG. Ceci permet de caler le ribosome sur le cadre de lecture du gène. Ce « balayage » de l'ARNm par le ribosome est facilité par l'activité hélicase de eIF4A qui, avec eIF4B, va ouvrir les structures secondaires éventuelles présentes dans le 5′-UTR. Une fois le codon de démarrage reconnu et l'interaction codon-anticodon formée, Il y a hydrolyse du GTP par eIF2, dissociation des facteurs de démarrage et recrutement de la sous-unité 60S. Le ribosome assemblé peut alors commencer la synthèse de la protéine.
Démarrage de la traduction dépendant des IRES
Les IRES (pour internal ribosome entry site) sont des régions structurées de la région 5′-UTR des ARNm qui permettent le recrutement direct du ribosome, indépendamment de la coiffe en 5′ et de certains des facteurs de démarrage de la traduction. Ces IRES sont principalement utilisés par des virus qui détournent la machinerie de traduction de la cellule infectée à leur profit. En général, le virus produit une enzyme capable d'inactiver un ou plusieurs des facteurs de démarrage classique, par exemple eIF4G, ce qui abolit la traduction de tous les ARNm cellulaires endogènes. L'ARNm viral utilise alors son IRES pour recruter le ribosome, ce qui lui permet d'être le seul à être traduit dans la cellule. Ce mécanisme a été identifié pour la première fois chez le poliovirus[15].
Les IRES correspondent à des régions de structure secondaire de l'ARNm, ce qui leur permet d'adopter un repliement tridimensionnel complexe. Cette structure complexe forme alors des interactions spécifiques avec certains facteurs de traduction ou directement avec le ribosome. Ceci permet de court-circuiter le mécanisme classique de recrutement du ribosome via la coiffe en 5′. Ainsi, par exemple, le virus de l'hépatite C possède un IRES dans sa région 5′-UTR qui recrute directement eIF3, sans nécessiter l'intervention de eIF4E et eIF4G. Il existe une grande variété d'IRES, qui permettent de se passer de plus ou moins de facteurs de démarrage cellulaires[16].
On a également rapporté l'existence d'IRES permettant l'expression de gènes cellulaires, comme c-myc, même si la fonctionnalité de ces IRES reste discutée[17].
Élongation
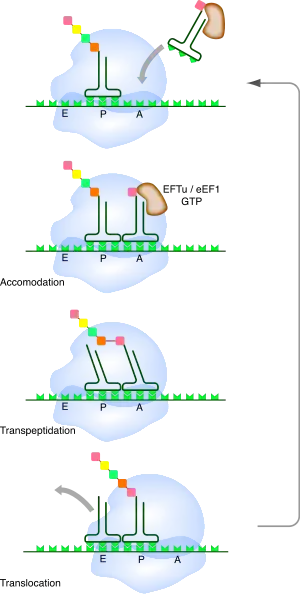
Au cours de la phase d'élongation, l'ARNm traverse le ribosome au niveau d'un sillon de la petite sous-unité. Pendant ce processus, un ou plusieurs ARN de transfert sont associés à l'ARNm au niveau de trois sites présents dans le ribosome :
- le site A (pour aminoacyl-ARNt) : il accueille les ARNt portant l'acide aminé qui va être ajouté à la chaîne. C'est au niveau du site A que s'effectue le décodage de l'information génétique, grâce à la reconnaissance de l'interaction entre le codon sur l'ARNm et l'anticodon sur l'ARNt ;
- le site P (pour peptidyl-ARNt) : il accueille l'ARNt qui porte la chaîne peptidique déjà synthétisée. Ce dernier est toujours apparié à l'ARNm par son anticodon ;
- le site E (pour l'anglais exit) ou site de sortie : il accueille l'ARNt « nu » après transfert de la chaîne peptidique sur l'ARNt suivant, juste avant qu'il ne quitte le ribosome.
Ces trois sites sont occupés séquentiellement par les ARNt au fur et à mesure de la progression du ribosome sur l'ARNm. À tout instant du processus de traduction, au plus deux de ces trois sites sont occupés simultanément : soit le site P et le site A, soit le site E et le site P.
Le début du cycle de synthèse démarre avec le ribosome ayant un seul ARNt fixé au site P par l'interaction codon-anticodon. Celui-ci porte estérifié à son extrémité 3′ le début de la chaîne peptidique synthétisée. Le site A est initialement vacant. Des ARNt aminoacylés diffusent dans le ribosome de manière stochastique. Ceux-ci sont associés à un facteur d'élongation (EF-Tu chez les bactéries, eEF1 chez les eucaryotes) qui est une GTPase. Le ribosome teste si l'appariement codon-anticodon est correct. C'est la petite sous-unité du ribosome qui effectue ce contrôle au niveau du site de décodage. Si l'appariement codon-anticodon n'est pas correct, l'ARNt est rejeté et le processus d'essai-erreur se répète. Lorsqu'un ARNt correct s'apparie enfin au codon, le facteur d'élongation hydrolyse une molécule de GTP, ce qui provoque un changement de conformation du complexe ribosomique et la dissociation du facteur d'élongation. Il y a alors formation de la liaison peptidique entre l'acide aminé porté par l'ARNt au site A et celui porté par l'ARNt au site P. Cette réaction se produit au niveau du site catalytique de la grande sous-unité du ribosome et conduit au transfert de la chaîne peptidique sur l'ARNt lié au site A. Le ribosome recrute alors un facteur de translocation (EF-G chez les bactéries, eEF2 chez les eucaryotes), lui aussi lié à une molécule de GTP. Ce facteur utilise l'énergie d'hydrolyse de ce GTP pour permettre la progression du ribosome sur l'ARNm. L'ARNt nu qui était au site P se retrouve décalé au site E de sortie et l'ARNt qui était au site A vient au site P. Le site A redevient ainsi vacant. L'ARNt nu du site E diffuse en dehors du ribosome et on revient dans l'état initial pour un nouveau cycle d'élongation.
ARN de transfert
Les ARN de transfert ou ARNt sont des molécules adaptatrices qui font l'intermédiaire entre le codon porté par l'ARN messager et l'acide aminé qui sera incorporé dans la protéine en cours de synthèse par le ribosome. À l'une de leurs extrémités se trouve une boucle contenant un triplet de nucléotides, l'anticodon, qui est complémentaire du codon. Cet appariement codon-anticodon permet la reconnaissance de l'ARNt par le ribosome. Du côté opposé à l'anticodon, les ARNt portent un acide aminé attaché par une liaison ester à leur extrémité 3′-OH. Chaque cellule contient un ensemble d'ARNt différents, capables de s'apparier aux différents codons et spécifiques chacun d'un acide aminé donné. Leurs structures sont analogues, ils adoptent une structure secondaire en forme de feuille de trèfle qui se replie en trois dimensions pour former une sorte de « L », avec l'anticodon d'un côté et l'acide aminé de l'autre.
Recodage
Le recodage est un évènement programmé, survenant pendant la lecture de l'ARNm par le ribosome et qui modifie le code initial[18]. La traduction en protéine résultante est donc différente de celle qu'on pourrait déduire à partir du code génétique standard. Il existe deux principaux évènements de recodage, avec des mécanismes distincts : le recodage du codon d'arrêt et le décalage du cadre de lecture.
Le recodage du codon d'arrêt permet à la cellule d'utiliser un codon de terminaison, en général le codon UGA, pour insérer un acide aminé spécifique. Ce mécanisme est en particulier utilisé pour insérer la sélénocystéine dans les sélénoprotéines. La présence du codon UGA ne suffit pas, il faut en plus la présence d'une structure secondaire spécifique sur l'ARNm et l'intervention de facteurs protéiques dédiés[19].
Le décalage du cadre de lecture est un mécanisme utilisé en particulier par les virus. Dans certaines conditions, le ribosome peut en effet « déraper » et glisser d'un nucléotide sur l'ARNm, ce qui permet de modifier la nature de la protéine synthétisée. Ce mécanisme est par exemple utilisé par le virus du SIDA pour produire les différentes enzymes nécessaires à sa réplication[20].
Ces décalages du cadre de lecture se produisent à des sites précis, au niveau d'une séquence « glissante », composée d'un nucléotide répété, comme AAAAAAA. Cette séquence est en général suivie d'une structure secondaire stable sur l'ARNm, souvent un pseudonœud[21]. Le ribosome est gêné dans sa progression par cette structure, ce qui provoque une pause dans la traduction. Pendant cette pause, il y a un codon AAA au site P du ribosome et un codon AAA au site A. Le ribosome en pause peut alors reculer d'un nucléotide en conservant ses deux ARNt fixés. Les appariements entre les codons et les anticodons (AAA / UUU) sont conservés, du fait de la nature particulière de la séquence glissante. La traduction continue alors sur une phase décalée. Ce glissement ne s'effectue pas à chaque traduction, mais avec une fréquence qui dépend de l'environnement et de la structure secondaire sur l'ARN messager. Ainsi le virus (ou la cellule) peut produire deux protéines distinctes à partir d'un seul ARNm.
Terminaison de la traduction
La terminaison de la traduction des protéines est le processus qui permet la libération de la protéine terminée une fois tout le gène lu par le ribosome, ainsi que le recyclage des différents acteurs impliqués, à savoir les sous-unités du ribosome, l'ARNm et le dernier ARNt[22]. Comme l'initiation et l'élongation, cette étape fait intervenir des protéines spécifiques, appelées facteurs de terminaison de la traduction (RF ou eRF, pour release factors). La terminaison est déclenchée par l'arrivée d'un codon d'arrêt dans le site A du ribosome. À ce stade, la protéine complète est encore accrochée par une liaison ester au dernier ARNt situé au site P. Il n'existe normalement pas d'ARNt portant un anticodon complémentaire du codon d'arrêt et pouvant se lier à l'ARNm au site A. À la place, un premier facteur de terminaison (RF1 ou RF2 chez les bactéries et eRF1 chez les eucaryotes) se lie au ribosome et interagit avec le codon d'arrêt. Ceci déclenche l'hydrolyse de la liaison ester entre la protéine et le dernier ARNt. La protéine ainsi libérée quitte le ribosome. Un second facteur (RF3 ou eRF3) qui est une GTPase se lie alors au ribosome, l'hydrolyse du GTP permettant alors le départ des deux facteurs. L'ARNt et le l'ARNm sont finalement libérés par l'action de RRF (ribosome release factor) et de facteurs d'élongation et d'initiation (EF-G et IF3 chez les bactéries).
Régulation de la traduction
Le contrôle de l'expression des gènes peut se faire au niveau de la traduction de l'ARN en protéine. Ce type de régulation agit principalement au niveau de l'étape de démarrage de la traduction par le ribosome. Plus rarement, c'est au niveau de la phase d'élongation ou de terminaison de la traduction qu'on peut avoir un contrôle. En modulant l'efficacité du recrutement du ribosome au niveau du codon de démarrage, la cellule contrôle la quantité de protéine qui est produite à partir de l'ARNm.
Dans de nombreux cas, ces mécanismes impliquent des structures spécifiques sur l'ARNm dont la traduction est régulée. La régulation peut faire intervenir un régulateur traductionnel exogène qui peut être soit une protéine, soit un autre ARN qui interfère avec la traduction par le ribosome en réponse à un signal extérieur (concentration d'un métabolite, température, présence d'un facteur protéique…). Ce régulateur peut être un activateur ou un répresseur, comme dans le cas de la régulation de la transcription.
La régulation de la traduction se passe dans le cytoplasme de la cellule, où se déroule cette étape de l'expression des gènes, tandis que la régulation de la transcription a lieu dans le noyau (chez les cellules eucaryotes).
Fidélité de la traduction
Le processus de traduction de l'ARNm en protéine a un taux d'erreur qui est de l'ordre de 10−4, soit un acide aminé incorrect sur 10000 acides aminés incorporés. La fidélité du mécanisme de traduction de l'ARNm par le ribosome repose sur deux étapes clés importantes. Il faut tout d'abord que chaque ARNt porte le bon acide aminé estérifié à son extrémité 3′, une étape qui est réalisée par une famille d'enzymes, les aminoacyl-ARNt synthétases. Les aminoacyl-ARNt synthétases ont souvent une fonction de relecture ou proofreading, comme les ADN polymérases, qui leur permet de vérifier que le produit de leur réaction est bien correct et sinon de l'hydrolyser. L'autre étape critique est la vérification de l'interaction codon-anticodon par le ribosome, une étape qui est aussi soumise à un processus de relecture, assisté par le facteur d'élongation de la traduction EF-Tu.
Contrôle qualité des ARNm
Les cellules vivantes ont développé différents mécanismes de contrôle qualité ou surveillance des ARNm. Cela permet suivant les cas de vérifier que l'ARNm est intact et qu'il code bien une protéine complète. Ceci évite la production de protéines anormales qui en s'accumulant pourraient finir par tuer la cellule. Ces mécanismes sont différents chez les bactéries et chez les eucaryotes.
Chez les eucaryotes, le principal mécanisme de contrôle qualité est la vérification de la présence de la coiffe et de la queue poly(A). Sans ces éléments, l'ARNm n'est pas exporté du noyau et ne peut être traduit car il n'y a pas formation du pseudo-cercle via l'interaction eIF4E-eIF4G-PABP décrite plus haut. Ainsi, par exemple, si un ARNm est endommagé et subit une coupure chimique ou enzymatique, il ne sera pas traduit, car l'extrémité 5′ qui porte la coiffe et l'extrémité 3′ qui porte la queue poly(A) seront séparées. Les eucaryotes disposent d'un autre mécanisme de contrôle qualité, le nonsense-mediated mRNA decay (« dégradation de l'ARNm par l'intermédiaire du [codon] non-sens »)[23]. Ce mécanisme conduit à la dégradation des ARNm qui contiennent un codon d'arrêt (dit également codon non-sens) prématuré, résultant par exemple d'une mutation dans le génome. C'est le ribosome, lors de la première traduction de l'ARNm, qui semble être le médiateur de ce contrôle.
Chez les bactéries, il existe un mécanisme de contrôle qualité qui permet de débloquer les ribosomes qui sont bloqués sur un ARNm, par exemple lorsqu'il ne contient pas de codon d'arrêt. Ceci peut se produire si l'ARNm a été clivé et qu'il manque la partie 3′ qui contient la fin du cadre ouvert de lecture et le codon d'arrêt. Ce mécanisme fait intervenir un ARN spécifique, l'ARNtm qui permet de débloquer le ribosome, par un mécanisme appelé trans-traduction qui fait changer le ribosome de message en cours de synthèse. La protéine incomplète est ainsi étiquetée par un peptide qui provoque sa dégradation rapide dans la cellule et évite donc l'accumulation de protéines aberrantes.
Dégradation
La dégradation des ARN messagers est effectuée par un complexe protéique appelé exosome, présent dans toutes les cellules eucaryotes et les archées. La dégradation est en général précédée par le raccourcissement de la queue poly(A) et par le retrait de la coiffe par une enzyme spécifique (decapping enzyme). Le dégradosome agit ensuite de manière exonucléolytique, c'est-à-dire qu'il hydrolyse progressivement l'ARN à partir d'une de ses extrémités, en l'occurrence à partir de l'extrémité 3′.
Chez les bactéries, il existe également un complexe spécifique, de nature différente, appelé dégradosome, chargé de recycler les ARN messagers.
Historique
Cette classe d'ARN a été isolée et étudiée pour la première fois en 1956 et appelée DNA-like RNA (ARN similaire à l'ADN) par Volkin et Astrachan[24], mais sans que ceux-ci n'en comprennent le rôle biologique. C'est François Gros qui a réellement caractérisé ces ARN quelques années plus tard[25] à la suite de l'hypothèse de leur existence formulée par Jacques Monod et François Jacob[26].
Les premières recherches dans les années 1960 portent sur des bactéries, avant de s'orienter ans les années 1970 vers des organismes plus complexes. Mary Edmonds et Aaron Shaktin découvrent alors les structures qui finissent et coiffent les ARN messagers et jouent un rôle essentiel pour les protéger et les lier aux ribosomes.
En 1993 et 1989, Phillip Sharp et Thomas Cech obtiennent un prix Nobel pour leur recherche sur la spécification de l'ARN et la synthèse des protéines[27].
Notes et références
- « Découverte de l’ARN messager, en 1961 », sur Institut Pasteur, (consulté le )
- Messenger RNA: origins of a discovery, Commentaire publié dans Nature par Alvin M. Weinberg
- (en) S. Brenner, F. Jacob et M. Meselson, « An unstable intermediate carrying information from genes to ribosomes for protein synthesis. », Nature, vol. 190, , p. 576-581
- Pour une hypothèse explicative de cette différence, voir l'article sur l'uracile
- (en) A.K. Banerjee, « 5'-terminal cap structure in eucaryotic messenger ribonucleic acids », Microbiol. Rev, vol. 44, no 2, , p. 175-205 (PMID 6247631, lire en ligne)
- (en) M. Edmonds, « A history of poly A sequences: from formation to factors to function. », Prog. Nucleic Acid Res. Mol. Biol., vol. 71, , p. 285-389 (PMID 12102557)
- (en) C. Saguez, J.R. Olesen et T.H. Jensen, « Formation of export-competent mRNP: escaping nuclear destruction », Curr. Opin. Cell. Biol., vol. 17, , p. 287-293 (PMID 15901499, DOI 10.1016/j.ceb.2005.04.009)
- (en) R. Benne, J. Van den Burg, J.P. Brakenhoff, PR. Sloof, J.H. Van Boom et M.C. Tromp, « Major transcript of the frameshifted coxII gene from trypanosome mitochondria contains four nucleotides that are not encoded in the DNA. », Cell, vol. 46, , p. 819-826 (PMID 3019552)
- (en) C. Job et J. Eberwine, « Localization and translation of mRNA in dendrites and axons. », Nat. Rev. Neurosci., vol. 2, , p. 889-898 (PMID 11733796)
- (en) M. Kozak, « Initiation of translation in prokaryotes and eukaryotes. », Gene, vol. 234, , p. 187-208 (PMID 10395892)
- (en) S.Z. Tarun Jr. et A.B. Sachs, « Association of the yeast poly(A) tail binding protein with translation initiation factor eIF-4G. », EMBO J., vol. 15, , p. 7168-7177 (PMID 9003792)
- (en) J. Shine et L. Dalgarno, « The 3'-terminal sequence of Escherichia coli 16S ribosomal RNA: complementarity to nonsense triplets and ribosome binding sites. », Proc. Natl. Acad. Sci. USA, vol. 71, , p. 1342-1346 (PMID 4598299)
- F. Poulin et S. Pyronnet, « Interactions moléculaires et initiation de la synthèse protéique », Médecine Sciences, vol. 16, , p. 617-622
- (en) M. Kozak, « Possible role of flanking nucleotides in recognition of the AUG initiator codon by eukaryotic ribosomes. », Nucleic acids res., vol. 9, , p. 5233-5252 (PMID 7301588)
- (en) J. Pelletier et N. Sonenberg, « Internal initiation of translation of eukaryotic mRNA directed by a sequence derived from poliovirus RNA. », Nature, vol. 334, , p. 320-325 (PMID 2839775)
- (en) L. Balvay, L. Soto Rifo, E.P. Ricci, D. Decimo et T. Ohlmann, « Structural and functional diversity of viral IRESes. », Biochim. Biophys. Acta, vol. 1789, , p. 542-557 (PMID 19632368)
- (en) M. Kozak, « A second look at cellular mRNA sequences said to function as internal ribosome entry sites. », Nucleic Acids Res., vol. 33, , p. 6593-6602 (PMID 16314320)
- (en) P.V. Baranov, R.F. Gesteland et J.F. Atkins, « Recoding: translational bifurcations in gene expression », Gene, vol. 286, , p. 187-201 (PMID 11943474, DOI 10.1016/S0378-1119(02)00423-7).
- (en) A. Böck, K. Forchhammer, J. Heider et C. Baron, « Selenoprotein synthesis: an expansion of the genetic code », Trends Biochem. Sci., vol. 16, , p. 463-467.
- (en) W. Wilson, M. Braddock, S.E. Adams, P.D. Rathjen, S.M. Kingsman et A.J. Kingsman, « HIV expression strategies: ribosomal frameshifting is directed by a short sequence in both mammalian and yeast systems », Cell, vol. 55, , p. 1159-1169 (PMID 3060262).
- (en) D.P. Giedroc, C.A. Theimer et P.L. Nixon, « Structure, stability and function of RNA pseudoknots involved in stimulating ribosomal frameshifting », J. Mol. Biol., vol. 298, , p. 167-185 (PMID 10764589).
- (en) L.L. Kisselev et R.H. Buckingham, « Translational termination comes of age », Trends Biochem. Sci., vol. 25, , p. 561-566 (PMID 11084369, DOI 10.1016/S0968-0004(00)01669-8)
- (en) Y.F. Chang, J.S. Imam et M.F. Wilkinson, « The nonsense-mediated decay RNA surveillance pathway. », Ann. Rev. Biochem., vol. 76, , p. 51-74 (PMID 17352659)
- (en) E. Volkin et L. Astrachan, « Intracellular distribution of labeled ribonucleic acid after phage infection of Escherichia coli. », Virology, vol. 2, , p. 433-437 (PMID 13352773)
- (en) F. Gros, H. Hiatt, W. Gilbert, C.G. Kurland, R.W. Risebrough et J.D. Watson, « Unstable ribonucleic acid revealed by pulse labelling of Escherichia coli », Nature, vol. 190, , p. 581-585 (PMID 13708983)
- (en) F. Jacob et J. Monod, « Genetic regulatory mechanisms in the synthesis of proteins. », J. Mol. Biol., vol. 3, , p. 318–356 (PMID 13718526)
- « 1960-1990: une archéologie de l’ARN messager », sur www.heidi.news (consulté le )
Articles connexes
Liens externes
- « Les joyaux de l’ARNm », La Méthode scientifique, France Culture, 15 septembre 2021.
- (en) Life of mRNA Flash animation
- (en) How mRNA is coded?: YouTube video
- Portail de la biologie cellulaire et moléculaire