Staphylococcus aureus résistant à la méticilline
Le Staphylococcus aureus résistant à la méticilline (SARM) ou, en anglais : Methicillin-resistant Staphylococcus aureus (MRSA) est un Staphylococcus aureus (couramment appelé « Staphylocoque doré ») caractérisé par sa résistance à un antibiotique, la méticilline (ou méthicilline).

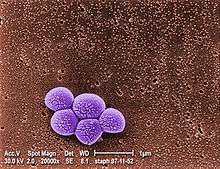
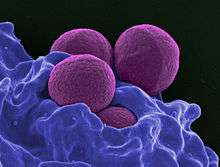
Microbiologie
Comme MSSA (pour Methicillin-sensitive Staphylococcus aureus - S. aureus sensible à la méticilline), le MRSA est un gram-positif, sphérique (coccus) bactérie qui est d'environ 1 micron de diamètre. Elle ne forme pas de spores et est non-mobile. Il forme des grappes ou des chaînes de raisin[1]. Contrairement à MSSA, le MRSA croît plus lentement sur divers médias et a été trouvé dans des colonies mixtes de MSSA. Le gène mecA, qui confère la résistance à un certain nombre d'antibiotiques, est présent dans le SARM et non dans le MSSA. Dans certains cas, le gène mecA est présent dans MSSA mais n'est pas exprimé. Le test de la réaction en chaîne par polymérase (PCR) est la méthode la plus précise pour identifier les souches de SARM. Des milieux de culture spécialisés ont été développés pour mieux différencier MSSA et MRSA et, dans certains cas, ils identifieront des souches spécifiques résistantes aux différents antibiotiques[1]. D'autres souches de S. aureus résistantes à l'oxacilline, la clindamycine, la teicoplanine et l'érythromycine ont également émergé. Ces souches résistantes peuvent ou non posséder le gène mecA. Des souches de S. aureus ont également développé une résistance à la vancomycine (VRSA). Une souche est seulement partiellement sensible à la vancomycine et s'appelle vancomycine-intermédiaire S. aureus (VISA). GISA est une souche de S. aureus présentant une sensibilité diminuée aux glycopeptides. S. aureus et est moins sensible à la vancomycine et à la teicoplanine. La résistance de S. aureus aux antibiotiques peut être quantifiée. Cela se fait en déterminant la quantité d'antibiotique en microgrammes / millilitre qui doit être utilisée pour inhiber la croissance. Si S. aureus est inhibé à une concentration de vancomycine inférieure ou égale à 4 microgrammes / millilitre, il est dit susceptible. Si une concentration de plus de 32 microgrammes / millilitre est nécessaire pour inhiber la croissance, on dit qu'elle est résistante[2].
Maladie émergente
Il a été découvert en 1961 en Grande-Bretagne[3], soit deux ans à peine après le début de l'utilisation de la méticilline. Les premières résistances étaient de type « hétérogène »[4], c'est-à-dire, identifiées uniquement après mise en culture à une température inférieure à 37°. Des souches résistantes ont ensuite été isolées également dans des pays n'ayant pas accès à cet antibiotique, probablement par acquisition de résistance croisée aux bêtalactamines naturelles[4]. Les premières souches américaines apparaissent dans les années 1970[4].
Il peut être identifié de manière classique par mise en culture en présence de méticilline (antibiogramme avec un résultat en 1 à 2 jours).
L'utilisation d'une méthode basée sur la réaction en chaîne par polymérase (PCR) permet d'avoir une identification en quelques heures, mais a une moindre spécificité[5].
Les infections à SARM sont généralement plus graves, avec une mortalité plus élevée[6].
Une nouvelle souche de SARM, d'origine bovine a été trouvée chez des malades d'hôpitaux irlandais, et par une autre équipe chez des patients du Royaume-Uni et du Danemark. Ce SARM est (septembre 2011) non détectable par les moyens habituels d'analyse. Un des auteurs de la découverte le Pr Coleman précise que c'est une nouvelle lignée de SARM, d'un type complètement différent des SARM découverts dans les années 1960, proche des SARM bovins, mais aussi résistant aux antibiotiques et de virulence semblable aux SARM humains nosocomiaux existants, risquant de produire de nombreuses nouvelles lignées par mutations[7],[8],[9].
Zone de prévalence
Le SARM, désormais courant en milieu hospitalier, était responsable de 37 % des cas fatals de septicémie au Royaume-Uni en 1999, soit 4 % de plus qu'en 1991. En 2003, il était responsable de près des deux-tiers des septicémies à staphylocoques aux États-Unis[10].
La moitié de tous les staphylocoques dorés aux États-Unis sont résistants à la pénicilline, la méticilline, la tétracycline et l'érythromycine. En 2005, les deux tiers des infections non hospitalières à staphylocoques concernent des souches résistantes à la méticilline[11].
Deux souches SARM hautement résistant aux antibiotiques qui n’étaient connues qu’aux États-Unis se sont répandues dans plusieurs villes colombiennes où elles causent des maladies graves, souvent mortelles[12].
Épidémiologie, écoépidémiologie


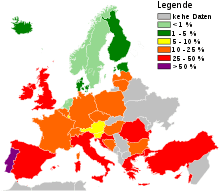
En Europe, l'AESA[14] estime en 2010 que les principaux réservoirs animaux du SARM (staphylocoques antibio-résistants) dans les pays touchés sont les cochons, veaux, et poulets de chair, dans les élevages industriels surtout.
Les infections sont parfois discrètes ou tout à fait asymptomatiques. Parmi les lignées de SARM, La souche « CC398 » est la plus souvent associée à un portage asymptomatique dans les élevages intensifs d'animaux producteurs de denrées alimentaires[14].
Le CC398 a été associée dans quelques cas à des infections profondes de la peau et des tissus mous, des pneumonies ou septicémies chez l'homme. Là où le CC398 est fréquent chez des animaux producteurs de denrées alimentaires, les éleveurs, vétérinaires et leurs proches risquent plus la colonisation et l'infection que la population générale[14].
Le SARM est souvent porteur de gènes d'entérotoxine mais il n'y a eu avant 2010 qu'un seul rapport d'intoxication alimentaire identifiée comme due à SARM.
La souche CC398 a aussi été isolée chez les animaux de compagnie (chiens et chat) et des chevaux dans les fermes ayant du bétail infecté, avec des cas sporadiques chez « de nombreux autres animaux de compagnie ». Cependant, selon l'AESA, aucune étude n'a spécifiquement évalué les risques de transmission à l'homme à partir de petits animaux ou suite aux contacts avec des chevaux ou de la viande chevaline[14].
Les infections à SARM chez les animaux de compagnie sont de plus en plus fréquentes, et presque toujours, les souches responsables de l'infection de ces animaux sont les mêmes que celles fréquemment rencontrées dans les hôpitaux de la même région géographique. Les êtres humains sont donc susceptibles de propager des infections à SARM aux animaux de compagnie et ceux-ci peuvent être un réservoir de la maladie pour l'homme[14].
Les chevaux peuvent être colonisés et/ou infectés par le SARM à partir de l'homme ou via d'autres sources animales dans leur milieu (chiens, chats...). Il existe des rapports sporadiques de cas de maladie chez l'homme concernant des infections cutanées généralement mineures attribuables à une source équine[14].
La souche CC398 peut entrer dans l'abattoir et circuler via les animaux vivants ou de la viande crue. Sur la base des données disponibles en 2008/2009, même si cette souche peut faire partie de la microflore endémique de certains abattoirs, le risque d'infection pour les travailleurs des abattoirs et les personnes y manipulant la viande semble rester faible[14].
Conséquences
Ce germe n'entraîne pas d'infection spécifique.
Les infections par les SARM entraînent des séjours hospitaliers plus longs et un coût de traitement plus important[15].
La mortalité par septicémie semble n'être supérieure qu'en cas d'infection par staphylocoque doré résistant à la méticilline[16].
Traitement
La vancomycine est l'antibiotique qui reste efficace dans ce cas pour l'instant, même s'il existe des souches résistantes à ce dernier (SARV).
Une nouvelle classe d'antibiotiques, les oxazolidinones, est disponible depuis les années 1990 et la première application commerciale, le linézolide est comparable à la vancomycine pour son efficacité contre le SARM.
La lysocine E, nouvel antibiotique découvert en 2014 par une équipe de chercheurs japonais de l'Université de Tokyo[17], est efficace contre le SARM[18].
Le traitement par la phagothérapie, éprouvé depuis des décennies en Russie et dans d'autres pays[19], est en cours d'évaluation en occident[19].
Prévention
- Prévenir la diffusion des germes à autrui implique notamment l'hygiène (en particulier des mains) et la mise en isolement de la personne infectée, même si cette dernière mesure repose plus sur de l'empirisme que sur une efficacité réellement démontrée[20].
- L'utilisation raisonnée et limitée des antibiotiques permet également de réduire la prévalence de ces germes[21].
- Selon l'AESA et son groupe d'experts sur les risques biologiques[14], les 4 phases d'interaction de la bactérie avec son environnement (y compris non-humain ou humain porteur sain) devraient être mieux suivies et comprises (infection, transport, colonisation et contamination).
L'usage raisonné des antibiotiques concerne donc aussi l'activité vétérinaire et surtout les élevage porcins[14].
Selon l'Autorité européenne de sécurité des aliments (AESA) (juin 2010), des aliments peuvent être contaminés et contaminants pour cette maladie. Il faudrait mieux surveiller les exploitations de porcs et le transport des porcs au sein de l'Union européenne, et notamment les porcheries industrielles qui présentent plus de risque en raison de leurs grandes tailles[14].
Dépistage
Le dépistage systématique des SARM chez certains patients est préconisé afin de tenter de diminuer le taux d'infection à ce germe. La présence de ce staphylocoque dans la sphère ORL (nez le plus fréquemment[22], gorge, oreilles) peut conduire à une décontamination et/ou à un isolement du patient porteur. L'intérêt de cette attitude n'est cependant pas formellement étayé[23],[24].
Les pays scandinaves préconisent le dépistage et le traitement systématique des porteurs, aussi bien parmi les patients que parmi le personnel soignant. Les raisons de la très faible prévalence du SARM dans ces pays ne sont pas clairement établies. On ignore si cela est dû à cette politique hospitalière agressive ou si cela provient d'autres causes[25].
Recherche scientifique
- Les herbes daphne genkwa et Olresin ont tous deux manifesté leur constitiuing que lorsqu'ils sont combinés avec des antibiotiques traditionnels suscitent une forte réponse contre les infections à SARM.[26]
- Divers extraits chimiques antibactériens provenant de diverses espèces de l'arbre Sweetgum (genre Liquidambar ont été étudiés pour leur activité dans l'inhibition du SARM. Plus précisément, ce sont: acide cinnamique, cinnamyl cinnamate, cinnamate d'éthyle, benzyl cinnamate, styrène, vanillin, cinnamyl alcohol , 2-phénylpropyl alcohol, 3-phenylpropyl cinnamate et vanillin[27].
- La livraison d'antibiotiques inhalés ainsi que l'administration systématique pour traiter le SARM sont en cours d'élaboration. On pense que cela améliorera les résultats de ceux atteints de fibrose kystique et d'autres infections respiratoires[28].
Notes et références
- (en) Patrick Murray, Manual of clinical microbiology, Washington, D.C, ASM Press, (ISBN 9781555813710) page 390
- (en) Washington Winn, Koneman's color atlas and textbook of diagnostic microbiology, Philadelphia, Lippincott Williams & Wilkins, (ISBN 0781730147) page 637
- (en) Jevons MP, « “Celbenin”-resistant staphylococci » BMJ. 1961;i:124-125.
- (en) Cookson B, « Five decades of MRSA: controversy and uncertainty continues » Lancet 2011;378:1291-1292.
- (en) De San N, Denis O, Gasasira MF, De Mendonca R, Nonhoff C, Struelens MJ, « Controlled evaluation of the IDI-MRSA assay for the detection of colonization by methicillin-resistant Staphylococcus aureus in diverse mucocutaneous specimens » J Clin Microbiol. 2007;45:1098-101.
- Cosgrove SE, Sakoulas G, Perencevich EN et Als. « Comparison of mortality associated with methicillin-resistant and methicillin-susceptible Staphylococcus aureus bacteremia: a meta-analysis » Clin Infect Dis. 2003;36:53-59.
- (en) Dick Ahlstrom, « New strain of MRSA superbug discovered in Dublin hospitals » Irish Times, 3 juin 2011.
- (en) « Trinity Scientists Identify A New Type of MRSA », Trinity College, Dublin 3 juin 2011.
- (en) Anna C. Shore, Emily C. Deasy, Peter Slickers, Grainne Brennan, Brian O'Connell, Stefan Monecke, Ralf Ehricht, David C. Coleman, « Detection of Staphylococcal Cassette Chromosome mec Type XI carrying Highly Divergent mecA, mecI, mecR1, blaZ and ccr Genes in Human Clinical Isolates and Clonal Complex 130 Methicillin-Resistant Staphylococcus aureus » Antimicrobial Agents and Chemotherapy 2011;55(8):3765-73. (Résumé).
- Klevens RM, Edwards JR, Tenover FC et Als. http://www.journals.uchicago.edu/CID/journal/issues/v42n3/38050/38050.html Changes in the epidemiology of methicillin-resistant Staphylococcus aureus in intensive care units in U.S. hospitals, 1992-2003], Clin Infect Dis, 2006;42:389-391.
- Klevens M, Morrison MA, Nadle J, Invasive methicillin-resistant staphylococcus aureus infections in the United States, JAMA, 2007;298:1763-1771.
- Arias C, MRSA USA300 Clone and VREF — A U.S.–Colombian connection?, N Eng J Med, 2008;359:2177-2179.
- Documentaire allemand (53 minutes) de Frank Bowinkelmann, intitulé en Français « Germes tueurs », réalisé pour la chaine Arte (en 2013) et, diffusé jeudi 9 janvier 2015 (22h20) puis rediffusé les 19.01.2015, 25.01.2015, 20.02.2015 et 20.02-27.02.2015 (lien provisoirement actif vers Arte +7).
- Assessment of the Public Health significance of meticillin resistant Staphylococcus aureus (MRSA) in animals and foods Question number: EFSA-Q-2008-300 Adopted: 5 March 2009 « Résumé »(Archive • Wikiwix • Archive.is • Google • Que faire ?) (consulté le 1er juin 2017) et « étude d'évaluation (Avis de l'EFSA) »(Archive • Wikiwix • Archive.is • Google • Que faire ?) (consulté le 1er juin 2017), 3 juin 2010.
- Engemann JJ, Carmeli Y, Cosgrove SE, Fowler VG, Bronstein MZ, Trivette SL et als. Adverse clinical and economic outcomes attributable to methicillin resistance among patients with Staphylococcus aureus surgical site infection, Clin Infect Dis, 2003;36:592-8.
- Laupland KB, Ross T, Gregson DB, Staphylococcus aureus bloodstream infections: risk factors, outcomes, and the influence of methicillin resistance in Calgary, Canada, 2000-2006, Clin Infect Dis, 2008;198:336-43.
- (en) Hiroshi Hamamoto et al., « Lysocin E is a new antibiotic that targets menaquinone in the bacterial membrane », Nature Chemical Biology, (DOI 10.1038/nchembio.1710, résumé).
- (en) Motoki Murai et al., « Total Synthesis and Biological Evaluation of the Antibiotic Lysocin E and Its Enantiomeric, Epimeric, and N-Demethylated Analogues », Angewandte Chemie International Edition, (DOI 10.1002/anie.201410270, résumé).
- Raphaëlle Maruchitch et Anuliina Savolainen, « Les phages, des virus guérisseurs. », sur Le Monde, .
- Cooper BS, Stone SP, Kibbler CC, Cookson BD, Roberts JA, Medley GF et als. Isolation measures in the hospital management of methicillin resistant Staphylococcus aureus (MRSA): systematic review of the literature, BMJ, 2004;329:533-40.
- Charbonneau P, Parienti JJ, Thibon P, Ramakers M, Daubin C, du Cheyron D et als. Fluoroquinolone use and methicillin-resistant Staphylococcus aureus isolation rates in hospitalized patients: a quasi experimental study, Clin Infect Dis, 2006;42:778-84.
- Kluytmans J, van Belkum A, Verbrugh H, Nasal carriage of Staphylococcus aureus: epidemiology, underlying mechanisms, and associated risks, Clin Microbiol Rev, 1997;10:505-20.
- Cooper BS, Stone SP, Kibbler CC, et als. Isolation measures in the hospital management of methicillin resistant Staphylococcus aureus (MRSA): systematic review of the literature, BMJ, 2004;329:533.
- Loeb M, Main C, Walker-Dilks C, Eady A, Antimicrobial drugs for treating meticillin resistant Staphylococcus aureus colonisation, Cochrane Database Syst Rev 2003;(3):CD003340.
- Kluytmans J, Struelens M, Meticillin resistant Staphylococcus aureus in the hospital, BMJ, 2009;338:b364.
- (en) Chiu-Fai Kuok, Sai-On Hoi, Chi-Fai Hoi, Chi-Hong Chan, Io-Hong Fong, Cheong-Kei Ngok, Li-Rong Meng et Pedro Fong, « Synergistic antibacterial effects of herbal extracts and antibiotics on methicillin-resistant Staphylococcus aureus: A computational and experimental study », Experimental Biology and Medicine (Maywood, N.J.), vol. 242, no 7, , p. 731–743 (ISSN 1535-3699, PMID 28118725, PMCID 5363687, DOI 10.1177/1535370216689828)
- (en) PhilipG Crandall, JodyM Lingbeck, CorlissA O′Bryan, ElizabethM Martin et JoshuaP Adams, « Sweetgum: An ancient source of beneficial compounds with modern benefits », Pharmacognosy Reviews, vol. 9, no 17, , p. 1 (ISSN 0973-7847, DOI 10.4103/0973-7847.156307)
- (en) Diego Maselli, Holly Keyt et Marcos Restrepo, « Inhaled Antibiotic Therapy in Chronic Respiratory Diseases (Review) », International Journal of Molecular Sciences, vol. 18, no 5, , p. 1062 (ISSN 1422-0067, DOI 10.3390/ijms18051062)
Voir aussi
- Staphylococcus
- Maladie infectieuse
- Maladie émergente
- Résistance aux antibiotiques
- Infection nosocomiale
- Épidémiologie
- Éco-épidémiologie
- Zoonose
- Portail de la médecine
- Portail de la microbiologie