Cytochrome c
Le cytochrome c est une petite hémoprotéine contenant une centaine de résidus d'acides aminés pour une masse d'environ 12 kDa. Il est très soluble dans l'eau, à raison d'environ 100 g·L-1 aux conditions physiologiques, ce qui le distingue des autres cytochromes, qui sont plutôt liposolubles.
| Cytochrome c | ||
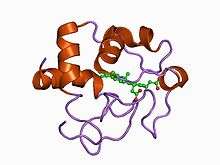
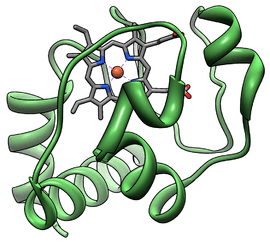 Structure du cytochrome c d'une cellule du myocarde de cheval montrant l'hème coordonné à un cation de fer (PDB 1HRC[1]) | ||
| Caractéristiques générales | ||
|---|---|---|
| Nom approuvé | Cytochrome c somatique | |
| Symbole | CYCS | |
| Synonymes | CYC, THC4, HCS | |
| Fonction | Respiration cellulaire | |
| Homo sapiens | ||
| Locus | 7p15.3 | |
| Masse moléculaire | 11 749 Da[2] | |
| Nombre de résidus | 105 acides aminés[2] | |
| Entrez | 54205 | |
| HUGO | 19986 | |
| OMIM | 123970 | |
| UniProt | P99999 | |
| RefSeq (ARNm) | NM_018947 | |
| RefSeq (protéine) | NP_061820.1 | |
| Ensembl | ENSG00000172115 | |
| PDB | 1J3S, 2N9I, 2N9J, 3NWV, 3ZCF, 3ZOO | |
|
GENATLAS • GeneTests • GoPubmed • HCOP • H-InvDB • Treefam • Vega | ||
Rôle dans la respiration cellulaire
Le cytochrome c intervient dans la respiration cellulaire au niveau de la chaîne respiratoire, où il transporte un électron par molécule. Il est, pour ce faire, associé à la membrane mitochondriale interne et assure le transfert des électrons entre la coenzyme Q-cytochrome c réductase (complexe III) et la cytochrome c oxydase (complexe IV). Chez l'homme, il est codé par le gène CYCS sur le chromosome 7[3].
Il peut également catalyser plusieurs réactions telles que des hydroxylations et l'oxydation aromatique, et présente également une activité peroxydase à travers l'oxydation de divers donneurs d'électrons tels que l'ABTS, l'acide 2-céto-4-thiométhylbutyrique et la 4-aminoantipyrine. Il intervient également dans au moins une forme de nitrite réductase[4].
Rôle dans le cycle cellulaire
Le cytochrome c peut également prendre une part active dans l'apoptose[5]. L'activation de la voie apoptotique mitochondriale a pour effet de faire apparaître des mégapores permettant le passage du cytochrome c dans le cytosol, où il se lie au facteur activateur des protéases apoptotiques (APAF1). Ceci déclenche une cascade de réactions aboutissant à l'activation de caspases, enzymes protéolytiques directement responsables du phénomène d'apoptose, et à la formation de l'apoptosome.
Origine et évolution
Le cytochrome c est une protéine hautement conservée à travers l'ensemble des espèces, et notamment chez les eucaryotes, où les différences se limitent à quelques résidus seulement. Tous les cytochromes c étudiés présentent un potentiel standard de +0,25 V, et on peut par exemple montrer que la cytochrome c oxydase humaine réagit avec le cytochrome c de blé. La séquence du cytochrome c chez l'homme est identique à celle du chimpanzé mais différente, par exemple, de celle du cheval[6]. Sa très large distribution chez les animaux, les plantes et de nombreux unicellulaires ainsi que sa petite taille (12 kDa) en font un outil de choix pour les études cladistiques visant à déterminer les parentés phylogénétiques entre espèces dans le cadre de la biologie de l'évolution[7].
Notes et références
- (en) Gordon W. Bushnell, Gordon V. Louie et Gary D. Brayer, « High-resolution three-dimensional structure of horse heart cytochrome c », Journal of Molecular Biology, vol. 214, no 2, , p. 585-595 (PMID 2166170, DOI 10.1016/0022-2836(90)90200-6, lire en ligne)
- Les valeurs de la masse et du nombre de résidus indiquées ici sont celles du précurseur protéique issu de la traduction du gène, avant modifications post-traductionnelles, et peuvent différer significativement des valeurs correspondantes pour la protéine fonctionnelle.
- (en) Marco Tafani, Natalie O. Karpinich, Kathryn A. Hurster, John G. Pastorino, Timothy Schneider, Matteo A. Russo et John L. Farber, « Cytochrome c Release upon Fas Receptor Activation Depends on Translocation of Full-length Bid and the Induction of the Mitochondrial Permeability Transition », Journal of Biological Chemistry, vol. 277, no 12, , p. 10073-10082 (PMID 11790791, DOI 10.1074/jbc.M111350200, lire en ligne)
- (en) Jörg Simon et Peter M. H. Kroneck, « The Production of Ammonia by Multiheme Cytochromes c », Metal Ions in Life Sciences, vol. 14, , p. 211-236 (PMID 25416396, DOI 10.1007/978-94-017-9269-1_9, lire en ligne)
- (en) Xuesong Liu, Caryn Naekyung Kim, Jie Yang, Ronald Jemmerson et Xiaodong Wang, « Induction of Apoptotic Program in Cell-Free Extracts: Requirement for dATP and Cytochrome c », Cell, vol. 86, no 1, , p. 147-157 (PMID 8689682, DOI 10.1016/S0092-8674(00)80085-9, lire en ligne)
- (en) Université de l'Indiana à Bloomington, « Cytochrome C Comparison Lab » [PDF] (consulté le 20 décembre 2015).
- (en) E. Margoliash, « Primary Structure and Evolution of Cytochrome C », Proceedings of the National Academy of Sciences of the United States of America, vol. 50, no 4, , p. 672-679 (PMID 14077496, PMCID 221244, DOI 10.1073/pnas.50.4.672, JSTOR 72215, Bibcode 1963PNAS...50..672M, lire en ligne)
- (en) K. Miki, S. Sogabe, A. Uno, T. Ezoe, N. Kasai, M. Saeda, Y. Matsuura et M. Miki, « Application of an automatic molecular-replacement procedure to crystal structure analysis of cytochrome c2 from Rhodopseudomonas viridis », Acta Crystallographica Section D – Structural Biology, vol. 50, , p. 271-275 (PMID 15299438, DOI 10.1107/S0907444993013952, lire en ligne)
- Portail de la biochimie
- Portail de la biologie cellulaire et moléculaire