U-Net
U-Net est un réseau de neurones à convolution développé pour la segmentation d'images biomédicales au département d'informatique de l'université de Fribourg en Allemagne[1]. Le réseau est basé sur le réseau entièrement convolutionnel[2] et son architecture a été modifiée et étendue pour fonctionner avec moins d’images d'entrainement et pour permettre une segmentation plus précise. La segmentation d'une image 512 * 512 prend moins d'une seconde sur un GPU récent.
Description
U-Net est une architecture plus élégante, appelée «réseau entièrement convolutionnel»[2].
L'idée principale est de compléter un contracting network par couches successives, les opérations de pooling sont remplacées par des opérateurs de suréchantillonnage. Par conséquent, ces couches augmentent la résolution de la sortie. De plus, une couche convolutionnelle successive peut alors apprendre à assembler une sortie précise à partir de cette information[1].
Une modification importante dans U-Net est qu'il existe un grand nombre de canaux de fonctions dans la partie de ré-échantillonnage, ce qui permet au réseau de propager des informations de contexte vers des couches de résolution supérieure. En conséquence, le chemin d'expansion est plus ou moins symétrique à la partie contractante et donne une architecture en forme de «U». Le réseau utilise uniquement la partie valide de chaque convolution sans aucune couche entièrement connectée[2]. Pour prédire les pixels dans la zone de bordure de l'image, le contexte manquant est extrapolé en reflétant l'image d'entrée. Cette stratégie de mosaïque est importante pour appliquer le réseau aux images volumineuses, car sinon la résolution serait limitée par la mémoire du processeur graphique.
Historique
U-Net a été créé par Olaf Ronneberger, Philipp Fischer et Thomas Brox en 2015 dans le cadre du document «UNet: réseaux de convolution pour la segmentation d'images biomédicales»[1].
C'est une amélioration et un développement de la FCN: Evan Shelhamer, Jonathan Long et Trevor Darrell (2014). "Fully convolutional networks for semantic segmentation"[2].
Architecture du réseau
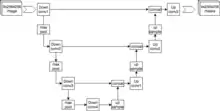
Le réseau se compose d'une partie contractante et une voie expansive, ce qui lui confère une architecture en forme de «U». La partie contractante est un réseau de convolution typique qui consiste en une application répétée de convolutions, chacune suivie d'une unité linéaire rectifiée (ReLU) et d'une opération de pooling maximum. Pendant la contraction, les informations spatiales sont réduites tandis que les informations sur les caractéristiques sont augmentées. La voie expansive combine les informations de caractéristiques géographiques et spatiales à travers une séquence de convolutions et concaténations ascendantes avec des fonctionnalités haute résolution issues de la voie contractante[3].
Applications
Il existe de nombreuses applications d' U-Net dans la segmentation d'images biomédicales, telles que la segmentation d'images cérébrales ('' BRATS ''[4]) et la segmentation d'images hépatiques ("siliver07"[5]). Voici quelques variantes et applications de U-Net comme suit:
(1). Régression par pixels avec U-Net et son application sur le "pansharpening"[6] ;
(2).3D U-Net: apprentissage de la segmentation volumétrique dense à partir d'une annotation fragmentée[7] ;
(3). TernausNet: U-Net avec encodeur VGG11 pré-formé sur ImageNet pour la segmentation d'images[8]..
Implémentations
jakeret (2017): “Tensorflow Unet”[9]
Code source U-Net de la reconnaissance de formes et du traitement d'images du département d'informatique de l'université de Fribourg, Allemagne[10].
Les articles de base sur le système[1],[2],[7],[8] ont été cités respectivement 3693, 7049, 442 et 22 sur Google Scholar au [11].
Références
- (en) Auteur inconnu « U-Net: Convolutional Networks for Biomedical Image Segmentation », {{{year}}}.
- (en) Auteur inconnu « Fully convolutional networks for semantic segmentation », {{{year}}}.
- « U-Net code »
- « MICCAI BraTS 2017: Scope | Section for Biomedical Image Analysis (SBIA) | Perelman School of Medicine at the University of Pennsylvania », www.med.upenn.edu (consulté le )
- « SLIVER07 : Home », www.sliver07.org (consulté le )
- (en) « Pixel-wise regression using U-Net and its application on pansharpening », Neurocomputing, vol. 312, , p. 364–371 (ISSN 0925-2312, DOI 10.1016/j.neucom.2018.05.103, lire en ligne)
- (en) Auteur inconnu « 3D U-Net: Learning Dense Volumetric Segmentation from Sparse Annotation », {{{year}}}.
- (en) Auteur inconnu « TernausNet: U-Net with VGG11 Encoder Pre-Trained on ImageNet for Image Segmentation », {{{year}}}.
- Joel Akeret, Generic U-Net Tensorflow implementation for image segmentation: jakeret/tf_unet, (lire en ligne)
- « U-Net: Convolutional Networks for Biomedical Image Segmentation », lmb.informatik.uni-freiburg.de (consulté le )
- Google Scholar citation data
- Portail des neurosciences
- Portail de la médecine
- Portail de l’informatique